Introducción
El nitrato es una de las principales reservas de nitrógeno fijado en el ciclo global del N (Falkowski y Godfrey, 2008; Klotz y Stein, 2008). El nitrato puede reducirse a través de la desnitrificación a óxido nitroso o gas dinitrógeno, por lo que se pierde de los ecosistemas terrestres y acuáticos, o a través de la amonificación a amonio que puede ser retenido (Brandes et al., 2007; Klotz y Stein, 2008, 2010). En ambas vías, el nitrato se reduce a nitrito. En los desnitrificadores heterótrofos y en los oxidantes de amoníaco, el nitrito se reduce aún más por las reductasas de nitrito formadoras de NO: NirS o NirK. La reducción del nitrito a amonio es catalizada por la nitrito reductasa asimiladora (NirB/NirA) o respiratoria (NrfA) formadora de amoníaco (Klotz y Stein, 2008, 2010). Los quimiolitótrofos que oxidan el amoníaco y los metanótrofos también pueden formar NO mediante la oxidación de la hidroxilamina, otra vía potencial de pérdida de nitrógeno en los ecosistemas terrestres y acuáticos (Campbell et al., 2011). Varias Epsilonproteobacterias que son capaces de utilizar el nitrato para la respiración y/o como única fuente de nitrógeno para el crecimiento no codifican homólogos de ninguno de los dos tipos de nitrito reductasa, lo que sugiere que estos organismos poseen un mecanismo no reconocido para la reducción del nitrito a amonio.
La anotación del genoma indica que no sólo Nautilia profundicola, sino varias Campylobacter spp. patógenas entre las Epsilonproteobacterias pueden utilizar el nitrato como fuente de nitrógeno para el crecimiento y/o como aceptor terminal de electrones. N. profundicola es un miembro del linaje más profundo de las Epsilonproteobacterias (Campbell et al., 2006; Smith et al., 2008) y sus parientes sólo se han encontrado en entornos de respiraderos hidrotermales de aguas profundas. En cambio, las Campylobacter spp. suelen estar asociadas a huéspedes animales y su presencia en otros entornos está mal definida. Mientras que los genomas de N. profundicola, Campylobacter concisus, C. curvus y C. fetus contienen genes que codifican un complejo periplásmico de guanina dinucleótido de nitrato reductasa (NAP), no hay genes que codifiquen reductasas de nitrito formadoras de amonio o de NO. Además, se identificó un gen napC/cycB en el genoma de N. profundicola (Campbell et al., 2009), lo cual es inusual ya que el complejo NAP en las Epsilonproteobacterias suele carecer de una proteína NapC (Kern y Simon, 2009; Klotz y Stein, 2010; Simon y Klotz, 2013). Basándonos en la reconstrucción metabólica informada por el genoma (Campbell et al., 2009), hemos propuesto recientemente una nueva vía para la asimilación del nitrato (Figura 1) por la que el nitrito se reduce a hidroxilamina mediante un módulo redox hidroxilamina:ubiquinona (HURM, es decir, una hidroxilamina deshidrogenasa dependiente de la quinona), que denominamos vía inversa-HURM. En N. profundicola, se cree que el HURM consiste en un citocromo cM552 relacionado con NapC/NrfH (cycB, NAMH_0559) que media la transferencia de electrones entre la reserva de quinonas y una hidroxilamina oxidorreductasa periplásmica (HAO, NAMH_1280). La HURM, que opera en la dirección de avance y consiste en la hidroxilamina deshidrogenasa (EC:1.7.2.6; HaoA3) conectada a un transportador de electrones de la proteína citocromo c (c554 y/o cM552 codificados por cycA y cycB, respectivamente), vincula la oxidación de la hidroxilamina a la piscina de quinonas en las bacterias aerobias oxidantes de amoníaco (Klotz y Stein, 2008; Simon y Klotz, 2013). En las bacterias anaerobias oxidantes del amoníaco (anammox), el módulo redox está formado por la hidracina oxidorreductasa y un citocromo c aún no identificado (Jetten et al., 2009; Simon y Klotz, 2013). En la vía inversa de la HURM, la hidroxilamina generada a partir del nitrito en el periplasma se transporta al citoplasma y se reduce a amonio a través de una hidroxilamina reductasa, también conocida como proteína de cluster híbrido (Har/Hcp). Así, las reacciones de aceptación de electrones propuestas en la vía de HURM inversa son:
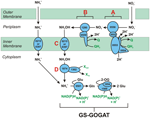
Figura 1. La vía inversa de HURM en N. profundicola. Los pasos individuales se señalan con letras mayúsculas en la figura. Los números en las proteínas representadas se refieren a las etiquetas de locus del genoma AmH (es decir, 1280 = NAMH_1280 = HaoA). (A) Reducción de nitrato por la nitrato reductasa periplásmica respiratoria (NapABGH, 0556-53), (B) HURM (Hydroxylamine:Ubiquinone Redox Module) compuesto por una hidroxilamina oxidorreductasa de funcionamiento inverso (HaoA, 1280) que funciona como una nitrito reductasa ocatahémica, que produce hidroxilamina utilizando electrones donados por un citocromo c tetrahémico de la familia NapC/NrfH/cM552 (CycB, 0559). (C) Transporte de hidroxilamina a través de transportadores de amoníaco relacionados con AmtB. (D) Reducción de la hidroxilamina a amonio por una proteína clúster híbrida/hidroxilamina reductasa (Hcp/Har, 1074) que utiliza el poder reductor de una proteína predicha que contiene Fe-S (1302) cuyo donante de electrones (X) es actualmente desconocido. La asimilación de amonio en la biomasa ocurre vía glutamina sintetasa y glutamina:2-oxoglutarato aminotransferasa (GS-GOGAT).
Los genomas de N. profundicola, C. concisus, C. curvus, C. fetus, y Caminibacter mediatlanticus cada uno contiene los genes que codifican las enzimas de la vía inversa-HURM mientras los genomas de otras Epsilonproteobacterias codifican los homólogos del clásico NirS/NirK formador de NO y siroheme asimilador NirA formador de amonio, o pentaheme respiratorio NrfA nitrito reductasas . Nuestra hipótesis es que la vía HURM inversa mejora la supervivencia y/o la dispersión de N. profundicola y Campylobacter spp. en entornos asociados o no asociados con el amonio. Sin embargo, para que esta hipótesis sea cierta, la vía HURM inversa debe proporcionar amonio para la biosíntesis y/o facilitar la conservación de la energía en estos organismos.

Tabla 1. Inventarios de genes del metabolismo del nitrógeno en las Epsilonproteobacterias secuenciadas por el genoma.
Resultados
Energética del crecimiento en N. profundicola
N. profundicola utiliza tanto el formiato como el hidrógeno como donantes de electrones para el metabolismo energético y se cultiva típicamente en polisulfuro como aceptor de electrones (Campbell et al., 2001; Smith et al., 2008). Los cálculos en condiciones estándar (1 M de todas las especies, 25°C, pH = 7) indican que cuando el nitrato se reduce por las reacciones de la vía inversa-HURM, la energía libre de Gibbs (Δ G°) es sustancialmente mayor que la disponible por la reducción del polisulfuro (Tabla 2). Por ejemplo, la Δ G° por mol de formiato oxidado con nitrato es 7,4 veces mayor que la del polisulfuro (-237 kJ mol-1 frente a -32 kJ mol-1). De este cálculo, uno puede predecir que el crecimiento de N. profundicola debe estimularse en las culturas con el nitrato como el aceptor del electrón en relación con el polisulfuro.
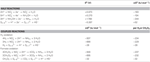
Table 2. Energías libres de reacción calculadas en condiciones estándar (1 M todas las especies, 25°C y pH = 7) para las semirreacciones de aceptación de electrones de compuestos de nitrógeno y polisulfuro y reacciones acopladas con formiato o hidrógeno como donante de electrones.
Crecimiento de N. profundicola con Nitrato
La caracterización anterior y el crecimiento de N. profundicola utilizaron medios con polisulfuro como una fuente combinada de azufre y aceptor de electrones (Campbell et al., 2001, 2009; Smith et al., 2008). Para aclarar aún más el papel del nitrato en el metabolismo energético de N. profundicola, la cepa AmH se cultivó con sulfuro como única fuente de azufre, de modo que el nitrato fue el único aceptor de electrones (Figura 2). El donante de electrones era una mezcla de hidrógeno y formiato en un medio sin otras fuentes de azufre o nitrógeno (Campbell et al., 2001). Las tasas de crecimiento con nitrato como aceptor potencial de electrones y fuente de nitrógeno y sulfuro (fuente de azufre) o polisulfuro (fuente de azufre y aceptor potencial de electrones) fueron más del doble de las tasas de crecimiento observadas en los cultivos con amonio como única fuente de nitrógeno y polisulfuro como aceptor de electrones y fuente de azufre. Como se esperaba, los cultivos de control negativo (sin aceptor de electrones) provistos de amonio como fuente de nitrógeno y sulfuro como fuente de azufre no pudieron crecer.
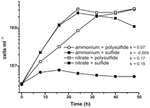
Figura 2. Crecimiento de N. profundicola con los aceptores de electrones, compuestos de nitrógeno y fuentes de azufre indicados. Las tasas de crecimiento se determinaron entre 8 y 24 h; k = constante de tasa de crecimiento específica (h-1). Cada punto es la media de 2 o 3 réplicas biológicas independientes.
Equilibrio de nitrógeno en los cultivos de N. profundicola
Para confirmar que el nivel de amonio producido por N. profundicola era superior a las necesidades biosintéticas, calculamos la demanda biosintética de nitrógeno como sigue. Asumiendo una relación C:N de 32:6.4 para el bacterioplancton de crecimiento exponencial (Vrede et al., 2002), 288 fg de C μm-3 para Escherichia coli de crecimiento exponencial (Loferer-Krossbacher et al., 1998), y un volumen celular de 0,021 μm3 calculado a partir de las dimensiones reportadas de N. profundicola (Smith et al., 2008), la producción de 1 × 108 células ml-1 requiere ~120 ng ml-1 de N (~9 μM), una cifra que no cambiaría independientemente de la fuente de N utilizada. N. profundicola no utilizó una cantidad significativa del amonio proporcionado durante el crecimiento con polisulfuro como aceptor de electrones (Figura 3, círculos abiertos), lo que concuerda con el bajo requerimiento de N calculado para producir las densidades celulares observadas. El cultivo con amonio más sulfuro no creció (Figura 2) y tampoco consumió amonio de forma detectable (Figura 3, círculos cerrados). En cambio, la cantidad de amonio producida por las células cultivadas con 5 mM de nitrato fue de 4,6 mM con polisulfuro y de 5,2 mM con sulfuro (Figura 3, cuadrados abiertos y cerrados, respectivamente). Si la vía HURM inversa en las Epsilonproteobacterias sirve principalmente para producir amonio para la biosíntesis, esperaríamos que niveles bajos de amonio exógeno reprimieran fuertemente la expresión génica de la vía HURM y la actividad de reducción de nitrato. Sin embargo, la conversión estequiométrica de nitrato a amonio en exceso de la necesidad de N predicha y el consumo de amonio observado en las culturas de amonio + polisulfuro es consistente con la hipótesis de que el nitrato se utiliza como un aceptor de electrones terminal por N. profundicola y que la amonificación de nitrato no está sujeta a la represión por el amonio.
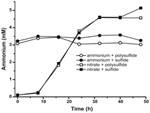
Figura 3. Las concentraciones del amonio midieron en las culturas de N. profundicola-con los aceptadores del electrón indicados, compuestos del nitrógeno, y fuentes del azufre. Cada punto es el promedio de 2 o 3 repeticiones biológicas independientes.
Incorporación de 15N-NO-3 en la Biomasa
El potencial para la asimilación de nitrato se evaluó examinando la incorporación de amonio o nitrato marcado con 15N en la biomasa de N. profundicola durante el crecimiento con todas las combinaciones anteriores de donantes de electrones y aceptores potenciales. N. profundicola creció en 20 Atom% de amonio marcado con 15N con polisulfuro produjo biomasa enriquecida a ~10 Atom% de 15N después de una ronda de cultivo por lotes (Figura 4), mientras que el control no marcado no se enriqueció en 15N. Los cultivos realizados con sulfuro y nitrato marcado con 20 Atom% de 15N produjeron biomasa enriquecida con 16 Atom% de 15N (Figura 4). La biomasa cultivada con nitrato contenía aproximadamente 5 veces más N que la biomasa cultivada con amonio (datos no mostrados), lo que puede explicar el enriquecimiento más extenso de 15N en las muestras cultivadas con nitrato.
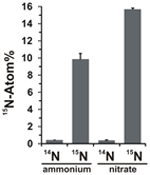
Figura 4. Captación de nitrógeno en la biomasa de N. profundicola determinada tras una ronda de cultivo por lotes con 5 mM de los compuestos de nitrógeno indicados suministrados en abundancia natural (14N) o en 20 Atom% 15N. Los valores son promedios de 3 réplicas biológicas independientes ± la desviación estándar.
Transcripción de Genes de la Vía Clave
La abundancia de la transcripción para los genes que codifican las enzimas de la vía inversa-HURM propuesta en N. profundicola se determinó por PCR cuantitativa en tiempo real en cultivos por lotes crecidos con amonio o nitrato. Todos los componentes de la vía propuesta mostraron un fuerte incremento en la abundancia de transcritos en los cultivos con nitrato (Figura 5). Los ARNm que codifican una subunidad de la nitrato reductasa (napA) fueron 4,6 veces (P < 0,05, prueba t heteroscedástica de dos colas) más abundantes en las células cultivadas con nitrato + sulfuro en relación con las células cultivadas con amonio + polisulfuro. Los transcritos de ambos componentes del HURM, haoA y cycB/napC, se incrementaron en 8,5 y 7,1 veces (P < 0,05 para ambos), respectivamente. En consonancia con nuestra predicción de que la hidroxilamina producida en el periplasma por el HURM sería transportada a través de los principales canales de proteínas facilitadoras, los ARNm que codifican ambos homólogos del transportador de amonio/metilamonio amtB se incrementaron en 4,5 veces (amtB-1, NAMH_0397, P < 0,08) y 10,3 veces (amtB-2, NAMH_0215, P > 0,10). La hidroxilamina es un potente mutágeno que debe ser desintoxicado al llegar al citoplasma. Proponemos que esto se lleva a cabo por Har/Hcp dependiente de NADH, basándonos en estudios del gen hcp en el grupo de genes de asimilación de nitrato de Rhodobacter capsulatus E1F1, donde se requiere para la reducción y asimilación de la hidroxilamina producida por una nitrato reductasa asimilatoria citoplasmática (Cabello et al., 2004; Pino et al., 2006). En consonancia con esta propuesta, el mayor cambio en la abundancia de transcripción se observó para har, que aumentó en 11,7 veces, pero este cambio no fue significativo (P > 0,10) debido a la alta variabilidad en las mediciones independientes de la abundancia de transcripción de har. Originalmente, habíamos propuesto que el donante de electrones de Har era una NADH deshidrogenasa codificada por NAMH_0542 (Campbell et al., 2009). Sin embargo, ahora creemos que un mejor candidato para esta función es NAMH_1302, una proteína predicha del clúster 4Fe-4S, basada en la conservación en otras Epsilonproteobacterias (Tabla 1). Consistente con esta predicción, las transcripciones de NAMH_1302 eran 4,6 veces más abundantes en las células cultivadas con nitrato en relación con el amonio, pero el cambio no era significativo (P > 0,10).
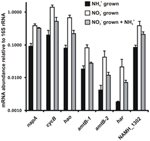
Figura 5. Niveles de ARNm en estado estable para los genes clave que codifican las enzimas de la vía de HURM inversa en N. profundicola cultivada con amonio + polisulfuro (barras negras), nitrato + sulfuro (barras blancas), o nitrato + polisulfuro después de la adición de amonio. La abundancia del transcrito se determinó mediante RT-PCR fluorescente y se normalizó con respecto a la abundancia del ARNr 16S determinada en cada muestra. Los valores son las medias de 3 réplicas biológicas independientes ± la desviación estándar.
Para comprobar la posible represión por amonio de la expresión de la vía, se añadió amonio a cultivos cultivados con nitrato + sulfuro y se cuantificó la abundancia de ARNm para los mismos genes. En general, la adición de amonio disminuyó la abundancia de ARNm de los genes que codifican la vía HURM inversa de 1,2 a 3,6 veces (Figura 5, barras grises), aunque este efecto sólo fue significativo (P < 0,10) para los transcritos haoA y napC/cycB que codifican las subunidades HURM. Curiosamente, la expresión de napA fue la que menos disminuyó (1,2 veces), lo que indica la necesidad de la respiración del nitrato en estas células. En ningún caso, la adición de amonio redujo los niveles de transcripción a los vistos en las culturas crecidas en amonio + polisulfuro.
Evidencia para un Intermedio de Hidroxilamina
Las mediciones directas de hidroxilamina en los filtrados de las culturas de N. profundicola crecidas con el nitrato encontraron niveles bajos, pero consistentemente detectables de hidroxilamina (5.7 ± 3.5 μM, SD, n = 3). Este nivel bajo es consistente con un intermedio cuya producción y tasas de consumo están estrechamente emparejadas.
Para determinar si la hidroxilamina podría ser metabolizada por N. profundicola, las suspensiones lavadas de células cultivadas con nitrato se incubaron en medio de crecimiento libre de nitrato con hidrógeno como el único donante de electrones. Se añadió hidroxilamina como aceptor potencial de electrones y se siguieron las concentraciones de hidroxilamina y amonio a lo largo del tiempo. Los resultados de este experimento indican que las células de N. profundicola cultivadas con nitrato convierten rápida y completamente la hidroxilamina en amonio (Figura 6). La tasa específica de conversión de hidroxilamina en este experimento fue de 28,6 μM min-1 108 células-1. La tasa de producción de amonio a partir de nitrato calculada para el crecimiento exponencial en la Figura 3 (8-24 h) es de ~3 μM min-1 (108 células)-1. Así, la tasa de conversión de hidroxilamina medida es muy superior a la tasa de producción de amonio observada durante el crecimiento de N. profundicola con nitrato. Un acoplamiento similar entre la producción y la eliminación de hidroxilamina se informó recientemente para la arquea quimiolitotrófica oxidante de amonio, Nitrosopumilus maritimus, donde la oxidación de hidroxilamina es un paso catabólico esencial (Vajrala et al., 2013).
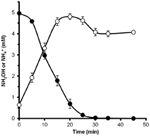
Figura 6. La hidroxilamina es rápidamente convertida en amonio por N. profundicola. Se siguieron las concentraciones de hidroxilamina (círculos cerrados) y amonio (círculos abiertos) tras la adición de 5 mM de hidroxilamina a una suspensión celular en reposo. Los puntos son los valores medios de tres réplicas ± la desviación estándar.
Discusión
Los datos presentados aquí son consistentes con una nueva vía para la reducción de nitrato en organismos que carecen de reductasas de nitrito clásicas formadoras de NO (NirS o NirK) o formadoras de amonio (NirB/NirA) con la cepa AmH de N. profundicola como un sistema modelo útil. Se ha propuesto una vía HURM inversa por la que el nitrito se reduce a hidroxilamina en el periplasma y posteriormente se reduce a amonio en el citoplasma (Campbell et al., 2009). La vía original se ha modificado ligeramente aquí para incluir un candidato más probable para el donante de electrones proximal a Har/Hcp, una proteína predicha que contiene 4Fe-4S codificada por NAMH_1302 (Figura 1). Los homólogos de NAMH_1302 se conservan en C. concisus (acceso GenBank YP_001466207), C. curvus (YP_001408944), C. fetus (YP_892678) y C. mediatlanticus (ZP_01871213) que poseen todos los demás genes de la vía inversa de HURM. Además, NAMH_1302 mostró un aumento en la abundancia de la transcripción en las células de N. profundicola cultivadas con nitrato en relación con el amonio.
Aunque la producción de hidroxilamina a partir de nitrato y nitrito tanto por vía química (Rollefson y Oldershaw, 1932) como biológica (Lindsey y Rhines, 1932) se conoce desde hace tiempo y se demostró que un complejo 3 reduce el nitrito a hidroxilamina cuando se le suministra un reductor (Kostera et al., 2010), la relación entre la hidroxilamina y la producción de amonio a partir de nitrito en la vía inversa de HURM aún no está clara. La captación de hidroxilamina extracelular por parte de las bacterias es conocida; la hidroxilamina suministrada de forma exógena se acumula fácilmente en el anammoxosoma de Kuenenia stuttgartiensis (Lindsay et al., 2001; Schmidt et al., 2004), un compartimento al que sólo se puede llegar atravesando las membranas de la célula y del anammoxosoma. La absorción propuesta de la hidroxilamina, altamente mutagénica, necesita protección contra las lesiones del ADN, lo que puede explicar en parte el amplio complemento de los sistemas de reparación del ADN identificados en N. profundicola (Campbell et al., 2009). La hidroxilamina también se ha propuesto como un intermediario de la producción de amonio a partir de nitrito en plantas y algas, ya que la nitrito reductasa dependiente de ferredoxina muestra una alta reactividad y especificidad para este sustrato (Kuznetsova et al., 2004; Hirasawa et al., 2010).
A diferencia de la función quinona reductasa de la HURM en los oxidantes de amoníaco (Klotz y Stein, 2008; Simon y Klotz, 2013), proponemos que la HURM en N. profundicola actúa, de forma similar a la NrfAH, como un sistema periplásmico de quinol oxidasa que transporta electrones desde la piscina de quinol para reducir el nitrito a hidroxilamina. Es probable que la vía inversa-HURM haya evolucionado a partir de un ancestro común del módulo de amonificación NrfAH y ONR en las bacterias anammox. Nuestra hipótesis es que esto ocurrió antes de que HURM -incluyendo un complejo trimérico HaoA unido covalentemente y capaz de desproporcionar la hidroxilamina- se utilizara como módulo catabólico en las bacterias anaerobias y aerobias que oxidan el amoníaco (Jetten et al., 2009; Klotz y Stein, 2010; Campbell et al., 2011; Kern et al., 2011; Simon y Klotz, 2013). Se ha discutido una inversión similar de la función de la actividad reductora a la oxidante efectuada por la formación de complejos dirigidos por enlaces covalentes para el citocromo c’-beta reductor de NO (cytS) y el citocromo P460 oxidante de NO (cytL) (Elmore et al., 2007). Los genes que codifican los módulos NrfAH y reverse-HURM coexisten en una sola secuencia del genoma bacteriano, C. fetus 82-40 (Tabla 1). Recientemente se ha demostrado que la hidroxilamina deshidrogenasa del citocromo c octahémico evolucionó como miembro de una superfamilia de proteínas multihémicas del citocromo c con funciones en los ciclos del nitrógeno y del azufre y que el citocromo cM552 (CycB), NrfH y NapC son miembros de otra superfamilia del citocromo c (Bergmann et al., 2005; Klotz y Stein, 2008; Kern et al., 2011; Simon y Klotz, 2013). También se sabe que los complejos NAP en Epsilonproteobacterias generalmente no contienen proteínas de subunidad NapC (Klotz et al., 2006; Sievert et al., 2008; Kern y Simon, 2009; Klotz y Stein, 2010; Simon y Klotz, 2013). La función inversa propuesta de un complejo n-cM552 (CycB/NapC) como complejo nitrito reductasa-quinol oxidasa es factible dada la presencia de ambas transcripciones y la ausencia del ligando crítico de la proteína tirosina en N. profundicola HaoA (Campbell et al., 2009), que separan los complejos NrfA y ONR, reductores de óxido de N, de los complejos HAO, oxidantes de óxido de N (Bergmann et al., 2005; Klotz y Stein, 2008; Kern et al., 2011; Simon y Klotz, 2013).
Los datos de asimilación de 15N establecieron que N. profundicola incorpora nitrógeno nítrico en la biomasa durante el crecimiento cuando se proporciona como única fuente de nitrógeno. Sin embargo, el gran exceso de producción de amonio a partir de nitrato sobre las demandas calculadas para la biosíntesis nos lleva a concluir que la función primaria de la reducción de nitrato a amonio por N. profundicola es la de un aceptor de electrones respiratorio eficiente. Esta conclusión está apoyada por el aumento de las tasas de crecimiento y los rendimientos en los cultivos de N. profundicola con nitrato como único aceptor de electrones. Además, los datos de expresión génica coinciden con nuestra propuesta (Campbell et al., 2009) de que esto ocurre a través de la función concertada de Nap, HURM (3 y cM552) y Har/Hcp como una nueva vía de asimilación de N y conservación de energía: la vía inversa-HURM. Por último, los datos indican que N. profundicola tiene la capacidad de reducir rápidamente la hidroxilamina a amonio, lo que concuerda con la propuesta de la hidroxilamina libre como un intermediario en la vía HURM inversa. La alta tasa de captación de hidroxilamina observada probablemente explica por qué no se observa de forma consistente durante el crecimiento de los cultivos en nitrato. Las mediciones preliminares de la producción de amonio a partir de nitrato o nitrito en células en reposo son similares a la tasa de 3 μM min-1 (108 células)-1 calculada a partir de los cultivos, ~10 veces menor que la tasa de consumo de hidroxilamina observada. Dadas estas tasas, la hidroxilamina debería consumirse inmediatamente después de su producción en los cultivos que crecen con nitrato. Como la hidroxilamina es mutagénica, nosotros hipotetizamos que esto refleja la presión selectiva previa para mantener los niveles bajos de hidroxilamina durante el crecimiento.
Mientras el etiquetado de la biomasa se ha intentado con 15N-NH2OH en las culturas de lote de N. profundicola, todavía no ha tenido éxito (los datos no se muestran). Dado que la hidroxilamina en el cultivo por lotes debe suministrarse a bajas concentraciones en relación con el nitrato no etiquetado para evitar la toxicidad y que la gran mayoría del N de la hidroxilamina debe convertirse en amonio (>90% basado en la Figura 3), este resultado no es sorprendente. Los experimentos de etiquetado a corto plazo con concentraciones mucho más altas de hidroxilamina etiquetada como la única fuente de N pueden establecer definitivamente este punto.
En la conclusión, los datos presentados aquí son consistentes con un modelo donde N. profundicola reduce el nitrato vía un intermedio de hidroxilamina libre. Estos experimentos todavía no excluyen completamente la posibilidad que la actividad de HURM inversa propuesta en N. profundicola funciona como un nitrito reductasa formador de amonio que transfiere 6 electrones sin liberar un intermedio de N-óxido. Los resultados no publicados indican que unas pocas mutaciones puntuales en el gen nrfA pueden convertir la citocromo c nitrito reductasa pentahémica en una enzima permeable que libera intermedios de óxido de N y no convierte estequiométricamente el nitrito en amonio (Jörg Simon, comuni. pers.). Dada la relación evolutiva entre nrfA y haoA (Bergmann et al., 2005; Simon y Klotz, 2013), el módulo reverso-HURM en N. profundicola podría funcionar naturalmente como un complejo enzimático productor de amonio e hidroxilamina, mientras que la actual NrfA opera «sin fugas» como una reductasa de nitrito productora de amonio. Otros experimentos utilizando inhibidores de Har/Hcp con nitrato o nitrito como sustrato en células enteras y/o estudios bioquímicos del complejo reverso-HURM para determinar el producto in vitro deberían proporcionar pruebas directas adicionales de esta hipótesis. Independientemente del mecanismo específico, la producción extensa de amonio muy por encima de las necesidades biosintéticas por N. profundicola sugiere que ella y las Epsilonproteobacterias relacionadas pueden servir un papel ecológico como una fuente de amonio en nitrato rico, amonio privado, ambientes hipóxicos donde ellos se encuentran comúnmente.
Materiales y Métodos
Cálculos de Energía Libre
Las energías libres de Gibbs (Tabla 2) se calcularon a partir de los potenciales redox estándar de las medias reacciones por la ecuación de Nernst (Δ G0 = -nFE0) o por la suma de las energías libres de las medias reacciones recogidas de la literatura (Thauer et al., 1977; van der Star et al., 2008). Todos los cálculos se realizaron en condiciones estándar (1 M todas las especies, 25°C, pH = 7).
Cultivo y análisis de compuestos de nitrógeno
Se cultivó la cepa AmH de N. profundicola (ATCC BAA-1463) y se contaron las células como se ha descrito previamente (Smith et al., 2008; Campbell et al., 2009). El amonio en los sobrenadantes del cultivo se cuantificó tras la derivación con cloruro de dansilo y la separación por HPLC con detección de fluorescencia (Shakila et al., 2001). El nitrato se cuantificó por HPLC con detección UV/Vis. Las concentraciones de hidroxilamina se determinaron en los filtrados de los cultivos (0,2 μm) según lo descrito por Frear y Burrell (1955).
Procedimientos de etiquetado con 15N
Se prepararon soluciones madre de nitrato 15N al 20 Atom% y de amonio 15N mezclando >99 Atom% y sales de abundancia natural para preparar el medio a una concentración final de 5 mM de nitrato o amonio según fuera necesario. Se cosecharon las células de los cultivos etiquetados con 15N mediante centrifugación y el pellet celular se lavó tres veces con agua nanopura para eliminar las sales. La suspensión celular se secó en cápsulas de estaño que se enviaron al laboratorio de isótopos estables de la Universidad de California-Davis, donde se determinó el % de átomos de 15N mediante espectrometría de masas de relación isotópica. Las muestras de control indicaron que no se había producido ninguna contaminación isotópica durante el procesamiento de las muestras (datos no mostrados).
PCR cuantitativa
Se utilizaron cebadores específicos de los genes (Tabla 3) para cuantificar la abundancia de ARNm en el ARN total extraído de N. profundicola como se describió previamente (Campbell et al., 2009).

Tabla 3. Primers y condiciones para la cuantificación Q-RT-PCR de genes específicos en muestras de ARN total extraídas de N. profundicola.
Captación de hidroxilamina
N. profundicola se cultivó con H2 y formiato como donantes de electrones y nitrato como único aceptor de electrones hasta la fase exponencial tardía. Las células se cosecharon por centrifugación. Todas las transferencias y lavados celulares se realizaron en una cámara anaeróbica de Coy Laboratories. Las células se resuspendieron con medio de crecimiento libre de nitratos hasta una concentración final de ~109 células ml-1 y se incubaron a 45°C durante 2 horas bajo una atmósfera de 80% de H2 + 20% de CO2 antes de añadir hidroxilamina (5 mM de concentración final) a partir de una solución madre preparada anaeróbicamente. Se filtraron alícuotas de la suspensión celular y se analizó la hidroxilamina como se ha descrito anteriormente y el amonio por HPLC después de la derivación con etoximetilenemalonato de dietilo (Gómez-Alonso et al., 2007).
Contribuciones de los autores
Thomas E. Hanson, Barbara J. Campbell y Martin G. Klotz diseñaron en colaboración los experimentos e interpretaron los resultados, Thomas E. Hanson realizó todos los análisis de compuestos nitrogenados y preparó las muestras para la espectrometría de masas, Barbara J. Campbell realizó todos los cultivos y llevó a cabo la PCR cuantitativa, Katie M. Kalis realizó los experimentos de captación de hidroxilamina, Mark A. Campbell identificó NAMH_1302 como gen relevante. Thomas E. Hanson redactó el artículo, que fue revisado posteriormente por Thomas E. Hanson, Barbara J. Campbell y Martin G. Klotz.
Declaración de conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un potencial conflicto de intereses.
Agradecimientos
Los autores desean agradecer a Lisa Y. Stein (Univ. Alberta-Edmonton), Jenn Macalady (Univ. Penn State), David Kirchman y Matt Cottrell (Univ. Delaware) por las lecturas críticas de los borradores del manuscrito, Jennifer Biddle por el acceso a una cámara anaeróbica y Kathy Coyne por el acceso a un HPLC. Este proyecto ha sido financiado con subvenciones de la National Science Foundation a Barbara J. Campbell y S. Craig Cary (EF-0333203), a Barbara J. Campbell (DEB-0640414), a Barbara J. Campbell y Thomas E. Hanson (MCB-0950691), y a Martin G. Klotz (MCB-0948202/1202648), por una subvención EPSCoR RII-2 a la Universidad de Delaware (EPS-0814251), y por fondos de incentivo proporcionados por la Universidad de Carolina del Norte a Martin G. Klotz.
Kern, M., y Simon, J. (2009). Cadenas de transporte de electrones y bioenergética del metabolismo respiratorio del nitrógeno en Wolinella succinogenes y otras Epsilonproteobacterias. Biochim. Biophys. Acta 1787, 646-656. doi: 10.1016/j.bbabio.2008.12.010
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Lindsey, G. A., y Rhines, C. M. (1932). La producción de hidroxilamina por la reducción de nitratos y nitritos por varios cultivos puros de bacterias. J. Bacteriol. 24, 489-492.
Pubmed Abstract | Pubmed Full Text
Loferer-Krossbacher, M., Klima, J., and Psenner, R. (1998). Determinación de la masa seca de las células bacterianas por microscopía electrónica de transmisión y análisis de imagen densitométrica. Appl. Environ. Microbiol. 64, 688-694.
Pubmed Abstract | Pubmed Full Text