Introduction
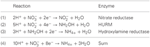
Nitrate is a major fixed nitrogen pool in the global N-cycle (Falkowski and Godfrey, 2008; Klotz and Stein, 2008). Azotan może być zredukowany poprzez denitryfikację do podtlenku azotu lub gazu dinitrogenowego, przez co jest tracony z ekosystemów lądowych i wodnych, lub poprzez amonifikację do amonu, który może być zatrzymany (Brandes i in., 2007; Klotz i Stein, 2008, 2010). W obu ścieżkach azotan jest redukowany do azotynu. W heterotroficznych denitryfikatorach i utleniaczach amoniaku, azotyn jest dalej redukowany przez reduktazy azotynowe tworzące NO: NirS lub NirK. Redukcja azotynów do amonu jest katalizowana przez asymilacyjną (NirB/NirA) lub respiracyjną (NrfA) reduktazę amonotwórczą azotynów (Klotz i Stein, 2008, 2010). Chemolitotrofy utleniające amoniak i metanotrofy mogą również tworzyć NO poprzez utlenianie hydroksyloaminy, co jest kolejną potencjalną drogą utraty azotu w ekosystemach lądowych i wodnych (Campbell i in., 2011). Kilka Epsilonproteobacteria, które są w stanie wykorzystać azotan do oddychania i/lub jako jedyne źródło azotu do wzrostu, nie koduje homologów żadnego z typów reduktazy azotynowej, co sugeruje, że organizmy te posiadają nierozpoznany mechanizm redukcji azotynów do amonu.
Anotacja genomu wskazuje, że nie tylko Nautilia profundicola, ale kilka patogennych Campylobacter spp. wśród Epsilonproteobacteria może wykorzystywać azotan jako źródło azotu do wzrostu i/lub jako terminalny akceptor elektronów. N. profundicola jest członkiem najgłębiej rozgałęzionej linii Epsilonproteobacteria (Campbell et al., 2006; Smith et al., 2008), a jej krewni zostali znalezieni tylko w głębinowych środowiskach ujść hydrotermalnych. W przeciwieństwie do nich, Campylobacter spp. są zwykle związane z żywicielami zwierzęcymi i ich obecność w innych środowiskach jest źle zdefiniowana. Podczas gdy genomy N. profundicola, Campylobacter concisus, C. curvus i C. fetus zawierają geny kodujące kompleks reduktazy azotanowej (NAP) związanej z peryplazmatycznym molibdopterynowym dinukleotydem guaniny, nie ma w nich genów kodujących reduktazy amonowe lub NO tworzące azotyny. Ponadto, w genomie N. profundicola zidentyfikowano gen napC/cycB (Campbell i in., 2009), co jest nietypowe, gdyż kompleks NAP u Epsilonproteobacteria zwykle pozbawiony jest białka NapC (Kern i Simon, 2009; Klotz i Stein, 2010; Simon i Klotz, 2013). W oparciu o rekonstrukcję metaboliczną opartą na genomie (Campbell i in., 2009), zaproponowaliśmy ostatnio nową ścieżkę asymilacji azotanów (Rysunek 1), w której azotyny są redukowane do hydroksyloaminy przez moduł redoks hydroksyloamina:ubichinon (HURM, tj. zależną od chinonu dehydrogenazę hydroksyloaminową), którą określamy jako ścieżkę odwrotną-HURM. U N. profundicola uważa się, że HURM składa się z cytochromu cM552 (cycB, NAMH_0559) związanego z NapC/NrfH, który pośredniczy w przenoszeniu elektronów między pulą chinonową a peryplazmatyczną oksydoreduktazą hydroksyloaminową (HAO, NAMH_1280). HURM, działaj±cy w kierunku do przodu i składaj±cy się z dehydrogenazy hydroksyloaminowej (EC:1.7.2.6; HaoA3) poł±czonej z białkowym transporterem elektronów cytochromu c (c554 i/lub cM552 kodowane odpowiednio przez cycA i cycB), ł±czy utlenianie hydroksyloaminy z pul± chinonow± u tlenowych bakterii utleniaj±cych amoniak (Klotz i Stein, 2008; Simon i Klotz, 2013). W beztlenowych bakteriach utleniających amoniak (anammox) moduł redoks składa się z oksydoreduktazy hydrazyny i niezidentyfikowanego jeszcze cytochromu c (Jetten i in., 2009; Simon i Klotz, 2013). W odwrotnym szlaku HURM hydroksyloamina generowana z azotynów w peryplazmie jest transportowana do cytoplazmy i redukowana do amonu przez reduktazę hydroksyloaminową, znaną również jako hybrydowe białko klasterowe (Har/Hcp). Zatem proponowane reakcje przyjmowania elektronów w szlaku odwrotnym-HURM to:
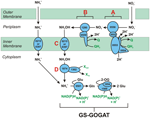
Rysunek 1. Szlak odwrotnego HURM u N. profundicola. Poszczególne etapy zaznaczone są na rysunku dużymi literami. Numery w przedstawionych białkach odnoszą się do znaczników locus z genomu AmH (tj. 1280 = NAMH_1280 = HaoA). (A) Redukcja azotanów przez oddechową peryplazmatyczną reduktazę azotanową (NapABGH, 0556-53), (B) HURM (Hydroxylamine:Ubiquinone Redox Module) składający się z odwrotnie działającej oksydoreduktazy hydroksyloaminy (HaoA, 1280) funkcjonującej jako reduktaza azotynów okataheme, która wytwarza hydroksyloaminę wykorzystując elektrony oddane przez tetra-hemowy cytochrom c z rodziny NapC/NrfH/cM552 (CycB, 0559). (C) Transport hydroksyloaminy przez transportery amoniaku związane z AmtB. (D) Redukcja hydroksyloaminy do amonu przez hybrydowe białko klasterowe/reduktazę hydroksyloaminy (Hcp/Har, 1074) wykorzystuj±ce siłę redukuj±c± z przewidywanego białka zawieraj±cego Fe-S (1302), którego donor elektronów (X) jest obecnie nieznany. Asymilacja amonu do biomasy zachodzi za pośrednictwem syntetazy glutaminy i aminotransferazy glutamina:2-oksoglutaran (GS-GOGAT).
Genomy N. profundicola, C. concisus, C. curvus, C. fetus, and Caminibacter mediatlanticus each contain genes encoding the enzymes of the reverse-HURM pathway while the genomes of other Epsilonproteobacteria encode homologues of the classical NO-forming NirS/NirK and ammonium-forming assimilatory siroheme NirA, or respiratory pentaheme NrfA nitrite reductases . Stawiamy hipotezę, że ścieżka odwrotna-HURM poprawia przeżywalność i/lub dyspersję N. profundicola i Campylobacter spp. w środowiskach, w których brakuje amonu, zarówno u gospodarzy, jak i w środowiskach niezwiązanych z gospodarzami. Jednakże, aby ta hipoteza była prawdziwa, ścieżka odwrotna-HURM musi dostarczać amonu do biosyntezy i/lub ułatwiać zachowanie energii u tych organizmów.

Tabela 1. Inwentarz genów metabolizmu azotu w genomie-sekwencjonowanych Epsilonproteobacteria.
Wyniki
Energetyka wzrostu u N. profundicola
N. profundicola wykorzystuje zarówno mrówczan jak i wodór jako donory elektronów do metabolizmu energetycznego i jest typowo hodowana na polisiarczku jako akceptorze elektronów (Campbell i in., 2001; Smith et al., 2008). Obliczenia w standardowych warunkach (1 M wszystkich gatunków, 25°C, pH = 7) wskazują, że kiedy azotan jest redukowany przez reakcje odwrotnej ścieżki HURM, wolna energia Gibbsa (Δ G°) jest znacznie większa niż ta dostępna z redukcji polisiarczku (Tabela 2). Na przykład, Δ G° na mol mrówczanu utlenionego azotanem jest 7,4-krotnie większa niż polisiarczku (-237 kJ mol-1 vs. -32 kJ mol-1). Na podstawie tych obliczeń można przewidywać, że wzrost N. profundicola powinien być stymulowany w hodowlach z azotanem jako akceptorem elektronów w stosunku do polisiarczku.
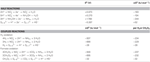
Tabela 2. Obliczone swobodne energie reakcji w warunkach standardowych (1 M wszystkich gatunków, 25°C i pH = 7) dla akceptujących elektrony półreakcji związków azotu i polisulfidu oraz reakcji sprzężonych z mrówczanem lub wodorem jako donorem elektronów.
Wzrost N. profundicola z azotanem
Pierwsza charakterystyka i wzrost N. profundicola wykorzystywały podłoża z polisiarczkiem jako połączonym źródłem siarki i akceptorem elektronów (Campbell i in., 2001, 2009; Smith i in., 2008). Aby dokładniej wyjaśnić rolę azotanu w metabolizmie energetycznym N. profundicola, szczep AmH hodowano z siarczkiem jako jedynym źródłem siarki, tak że azotan był jedynym akceptorem elektronów (Rysunek 2). Donorem elektronów była mieszanina wodoru i mrówczanu w podłożu nie zawierającym innych źródeł siarki ani azotu (Campbell i in., 2001). Szybkości wzrostu z azotanem jako potencjalnym akceptorem elektronów i źródłem azotu oraz siarczkiem (źródłem siarki) lub polisiarczkiem (źródłem siarki i potencjalnym akceptorem elektronów) były ponad dwukrotnie wyższe niż obserwowane w hodowlach wykorzystujących amon jako jedyne źródło azotu i polisiarczek jako akceptor elektronów i źródło siarki. Zgodnie z oczekiwaniami, negatywna kontrola (bez akceptora elektronów) kultur dostarczanych z amonem jako źródłem azotu i siarczkiem jako źródłem siarki nie była w stanie rosnąć.
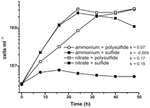
Rysunek 2. Wzrost N. profundicola przy użyciu wskazanych akceptorów elektronów, związków azotu i źródeł siarki. Szybkość wzrostu określono pomiędzy 8 a 24 godziną; k = stała szybkości wzrostu (h-1). Każdy punkt to średnia z 2 lub 3 niezależnych powtórzeń biologicznych.
Nitrogen Balance in N. profundicola Cultures
Aby potwierdzić, że poziom amonu produkowanego przez N. profundicola przekraczał potrzeby biosyntezy, obliczyliśmy biosyntetyczne zapotrzebowanie na azot w następujący sposób. Przyjmując stosunek C:N 32:6.4 dla wykładniczo rosnącego bakterioplanktonu (Vrede i in., 2002), 288 fg C μm-3 dla wykładniczo rosnącej Escherichia coli (Loferer-Krossbacher i in., 1998), i objętości komórek 0,021 μm3 obliczonej na podstawie podanych wymiarów N. profundicola (Smith i in., 2008), produkcja 1 × 108 komórek ml-1 wymaga ~120 ng ml-1 N (~9 μM), liczba ta nie zmieniłaby się niezależnie od wykorzystanego źródła N. N. profundicola nie wykorzystała znacznej ilości amonu dostarczonego podczas wzrostu z polisiarczkiem jako akceptorem elektronów (Rysunek 3, otwarte okręgi), co zgadza się z niskim zapotrzebowaniem na N obliczonym w celu wytworzenia obserwowanych gęstości komórek. Kultura amon plus siarczek nie rosła (rysunek 2) i również nie zużywała w sposób wykrywalny amonu (rysunek 3, zamknięte okręgi). Dla kontrastu, ilość amonu produkowanego przez komórki hodowane z 5 mM azotanu wynosiła 4,6 mM z polisiarczkiem i 5,2 mM z siarczkiem (Rysunek 3, odpowiednio otwarte i zamknięte kwadraty). Jeśli ścieżka odwrotna HURM w Epsilonproteobacteria służy przede wszystkim do produkcji amonu do biosyntezy, spodziewalibyśmy się, że niski poziom egzogennego amonu będzie silnie represjonował ekspresję genów ścieżki HURM i aktywność redukcji azotanów. Jednakże, stechiometryczna konwersja azotanu do amonu przekraczająca przewidywane zapotrzebowanie na N i obserwowane zużycie amonu w kulturach amon + polisiarczek jest zgodne z hipotezą, że azotan jest wykorzystywany jako terminalny akceptor elektronów przez N. profundicola i że amonifikacja azotanu nie podlega represji przez amon.
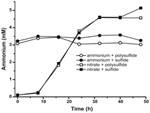
Rysunek 3. Stężenia amonu mierzone w kulturach N. profundicola zawierających wskazane akceptory elektronów, związki azotu i źródła siarki. Każdy punkt to średnia z 2 lub 3 niezależnych powtórzeń biologicznych.
Inkorporacja 15N-NO-3 do biomasy
Potencjał asymilacji azotanów oceniano badając inkorporację znakowanego 15N amonu lub azotanu do biomasy N. profundicola podczas wzrostu z wszystkimi powyższymi kombinacjami donorów elektronów i potencjalnych akceptorów. N. profundicola hodowana na 20 Atom% 15N znakowanym amonie z polisiarczkiem wytworzyła biomasę wzbogaconą do ~10 Atom% 15N po jednej rundzie hodowli wsadowej (Rysunek 4), podczas gdy kontrola bez znakowania nie była wzbogacona w 15N. Kultury hodowane z siarczkiem i 20 Atom% azotanem znakowanym 15N wytworzyły biomasę wzbogaconą do 16 Atom% 15N (Rysunek 4). Biomasa hodowana na azotanach zawierała około 5-krotnie więcej N niż biomasa hodowana na amonach (dane nie pokazane), co może tłumaczyć większe wzbogacenie 15N w próbkach hodowanych na azotanach.
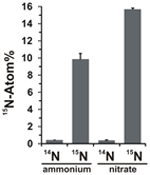
Rysunek 4. Pobór azotu do biomasy N. profundicola określony po jednej rundzie hodowli wsadowej z 5 mM wskazanych związków azotu dostarczonych w naturalnej obfitości (14N) lub w 20 Atom% 15N. Wartości są średnimi z 3 niezależnych powtórzeń biologicznych ± odchylenie standardowe.
Transskrypcja kluczowych genów szlaku
Objętość transkryptów dla genów kodujących enzymy proponowanego szlaku odwrotnego HURM w N. profundicola została określona przez ilościową PCR w czasie rzeczywistym w hodowlach wsadowych hodowanych z amonem lub azotanem. Wszystkie komponenty proponowanego szlaku wykazywały silny wzrost obfitości transkryptów w hodowlach azotanowych (Rysunek 5). mRNA kodujące podjednostkę reduktazy azotanowej (napA) było 4,6-krotnie (P < 0,05, dwusilnikowy heteroscedastyczny test t) bardziej obfite w komórkach hodowanych na azotan + siarczek w stosunku do komórek hodowanych na amon + wielosiarczek. Transkrypty obu składników HURM, haoA i cycB/napC, były zwiększone odpowiednio 8,5- i 7,1-krotnie (P < 0,05 dla obu). Zgodnie z naszymi przewidywaniami, że hydroksyloamina produkowana w peryplazmie przez HURM będzie transportowana przez główne kanały białek pośredniczących, mRNA kodujące oba homologi transportera amonu/metyloamonu amtB były zwiększone 4,5-krotnie (amtB-1, NAMH_0397, P < 0,08) i 10,3-krotnie (amtB-2, NAMH_0215, P > 0,10). Hydroksyloamina jest silnym mutagenem, który musi zostać zdetoksykowany po dostaniu się do cytoplazmy. Proponujemy, że jest to realizowane przez zależny od NADH Har/Hcp w oparciu o badania genu hcp w klastrze genów asymilacji azotanów Rhodobacter capsulatus E1F1, gdzie jest on wymagany do redukcji i asymilacji hydroksyloaminy wytwarzanej przez cytoplazmatyczną asymilacyjną reduktazę azotanową (Cabello i in., 2004; Pino i in., 2006). Zgodnie z tą propozycją, największą zmianę w liczebności transkryptu zaobserwowano dla har, która wzrosła 11,7-krotnie, ale zmiana ta nie była znacząca (P > 0,10) ze względu na dużą zmienność w niezależnych pomiarach liczebności transkryptu har. Pierwotnie zaproponowaliśmy, że donorem elektronów dla har jest dehydrogenaza NADH kodowana przez NAMH_0542 (Campbell i in., 2009). Jednak obecnie uważamy, że lepszym kandydatem do tej funkcji jest NAMH_1302, przewidywane białko klastra 4Fe-4S, na podstawie zachowania w innych Epsilonproteobacteria (Tabela 1). Zgodnie z tym przewidywaniem, transkrypty NAMH_1302 były 4,6-krotnie obfitsze w komórkach hodowanych na azotanie w stosunku do amonu, ale zmiana nie była znacząca (P > 0,10).
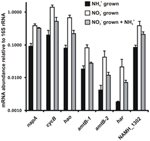
Figura 5. Poziomy mRNA w stanie ustalonym dla kluczowych genów kodujących enzymy odwrotnej ścieżki HURM u N. profundicola hodowanej z amonem + wielosiarczkiem (czarne słupki), azotanem + siarczkiem (białe słupki) lub azotanem + wielosiarczkiem po dodaniu amonu. Obfitość transkryptów oznaczono metodą fluorescencyjnej RT-PCR i znormalizowano do obfitości 16S rRNA oznaczonej w każdej próbce. Wartości są średnimi z 3 niezależnych powtórzeń biologicznych ± odchylenie standardowe.
Aby sprawdzić potencjalną represję amonu w ekspresji szlaku, dodano amon do kultur hodowanych na azotanie + siarczku i oznaczono ilościowo obfitość mRNA dla tych samych genów. Ogólnie rzecz biorąc, dodatek amonu zmniejszył obfitość mRNA genów kodujących szlak odwrotnej HURM od 1,2- do 3,6-krotnie (Rysunek 5, szare słupki), chociaż efekt ten był znaczący (P < 0,10) tylko dla transkryptów haoA i napC/cycB, które kodują podjednostki HURM. Co ciekawe, ekspresja napA zmniejszyła się najmniej (1,2-krotnie), wskazując na potrzebę oddychania azotanowego w tych komórkach. W żadnym przypadku dodatek amonu nie obniżył poziomu transkryptów do poziomu obserwowanego w kulturach hodowanych w amonie + polisiarczku.
Dowód na pośredni poziom hydroksyloaminy
Bezpośrednie pomiary hydroksyloaminy w filtratach kultur N. profundicola hodowanych z azotanem wykazały niski, ale stale wykrywalny poziom hydroksyloaminy (5,7 ± 3,5 μM, SD, n = 3). Aby określić, czy hydroksyloamina może być metabolizowana przez N. profundicola, wypłukane zawiesiny komórek hodowanych na azotanach inkubowano w pożywce bezazotanowej z wodorem jako jedynym donorem elektronów. Hydroksyloamina była dodawana jako potencjalny akceptor elektronów, a stężenia hydroksyloaminy i amonu były śledzone w czasie. Wyniki tego eksperymentu wskazują, że hodowane na azotanach komórki N. profundicola szybko i całkowicie przekształcają hydroksyloaminę do amonu (Rysunek 6). Specyficzna szybkość konwersji hydroksyloaminy w tym eksperymencie wynosiła 28.6 μM min-1 108 komórek-1. Szybkość produkcji amonu z azotanu obliczona dla wzrostu wykładniczego na rysunku 3 (8-24 h) wynosi ~3 μM min-1 (108 komórek)-1. Tak więc, zmierzona szybkość konwersji hydroksyloaminy znacznie przewyższa szybkość produkcji amonu obserwowaną podczas wzrostu N. profundicola z azotanem. Podobnie ścisłe sprzężenie między produkcją hydroksyloaminy a jej usuwaniem odnotowano niedawno u chemolitotroficznego archeona utleniającego amoniak, Nitrosopumilus maritimus, u którego utlenianie hydroksyloaminy jest istotnym etapem katabolicznym (Vajrala i in., 2013).
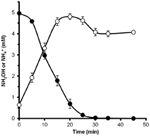
Rysunek 6. Hydroksyloamina jest szybko przekształcana do amonu przez N. profundicola. Stężenia hydroksyloaminy (zamknięte kółka) i amonu (otwarte kółka) śledzono po dodaniu 5 mM hydroksyloaminy do zawiesiny komórek w stanie spoczynku. Punkty są wartościami średnimi z trzech powtórzeń ± odchylenie standardowe.
Dyskusja
Przedstawione tutaj dane są zgodne z nową ścieżką redukcji azotanów w organizmach, w których brakuje klasycznych reduktaz azotynowych tworzących NO (NirS lub NirK) lub tworzących amon (NirB/NirA), z N. profundicola szczep AmH jako użytecznym systemem modelowym. Zaproponowano odwrotny szlak HURM, w którym azotyny są redukowane do hydroksyloaminy w peryplazmie, a następnie redukowane do amonu w cytoplazmie (Campbell i in., 2009). Oryginalny szlak został nieco zmodyfikowany, aby uwzględnić bardziej prawdopodobnego kandydata na proksymalnego donora elektronów dla Har/Hcp, przewidywane białko zawierające 4Fe-4S kodowane przez NAMH_1302 (Rysunek 1). Homologi NAMH_1302 są konserwowane u C. concisus (akcesja GenBank YP_001466207), C. curvus (YP_001408944), C. fetus (YP_892678) i C. mediatlanticus (ZP_01871213), które posiadają wszystkie pozostałe geny dla ścieżki odwrotnej HURM. Ponadto, NAMH_1302 wykazywał wzrost liczebności transkryptu w komórkach N. profundicola hodowanych na azotanie w stosunku do amonu.
Podczas gdy produkcja hydroksyloaminy z azotanu i azotynu zarówno drogą chemiczną (Rollefson i Oldershaw, 1932), jak i biologiczną (Lindsey i Rhines, 1932) jest znana od dawna i wykazano, że kompleks 3 redukuje azotyn do hydroksyloaminy po dostarczeniu reduktanta (Kostera i in., 2010), stosunek hydroksyloaminy do produkcji amonu z azotynów w odwrotnym szlaku HURM nie jest jeszcze jasny. Znany jest wychwyt zewnątrzkomórkowej hydroksyloaminy przez bakterie; egzogennie dostarczona hydroksyloamina łatwo gromadzi się w anammoksosomie Kuenenia stuttgartiensis (Lindsay i in., 2001; Schmidt i in., 2004), przedziale, do którego można dotrzeć jedynie poprzez przekroczenie błony komórkowej i anammoksosomowej. Proponowany pobór silnie mutagennej hydroksyloaminy wymaga ochrony przed uszkodzeniami DNA, co może częściowo tłumaczyć szeroki zestaw systemów naprawy DNA zidentyfikowanych u N. profundicola (Campbell i in., 2009). Hydroksyloamina została również zaproponowana jako substancja pośrednia w produkcji amonu z azotynów w roślinach i glonach, ponieważ zależna od ferredoksyny reduktaza azotynowa wykazuje wysoką reaktywność i specyficzność dla tego substratu (Kuznetsova i in., 2004; Hirasawa i in.., 2010).
W przeciwieństwie do funkcji reduktazy chinonowej HURM w amoniakalnych utleniaczach (Klotz i Stein, 2008; Simon i Klotz, 2013), proponujemy, że HURM w N. profundicola działa, podobnie jak NrfAH, jako peryplazmatyczny system oksydazy chinolowej, który przerzuca elektrony z puli chinolowej do redukcji azotynów do hydroksyloaminy. Jest prawdopodobne, że szlak odwrotny-HURM wyewoluował od wspólnego przodka NrfAH i modułu amonifikacyjnego ONR w bakteriach anammox. Hipotezujemy, że nastąpiło to zanim HURM – łącznie z kowalencyjnie związanym trimerycznym kompleksem HaoA zdolnym do dysproporcjonowania hydroksyloaminy – został wykorzystany jako moduł kataboliczny zarówno w beztlenowych jak i tlenowych bakteriach utleniających amoniak (Jetten i in., 2009; Klotz i Stein, 2010; Campbell i in., 2011; Kern i in., 2011; Simon i Klotz, 2013). Podobne odwrócenie funkcji z aktywności redukującej na utleniającą, dokonujące się poprzez tworzenie kompleksów z wiązaniami kowalencyjnymi, omówiono dla redukującego NO cytochromu c’-beta (cytS) i utleniającego NO cytochromu P460 (cytL) (Elmore i in., 2007). Geny kodujące moduły NrfAH i reverse-HURM współistnieją tylko w jednej sekwencji genomu bakteryjnego, C. fetus 82-40 (tab. 1). Ostatnio wykazano, że dehydrogenaza hydroksyloaminowa oktahemu cytochromu c wyewoluowała jako członek wielozasadowej nadrodziny białek cytochromu c z funkcjami w cyklach azotowym i siarkowym oraz że cytochrom cM552 (CycB), NrfH i NapC są członkami innej nadrodziny cytochromów c (Bergmann i in., 2005; Klotz i Stein, 2008; Kern i in., 2011; Simon i Klotz, 2013). Wiadomo również, że kompleksy NAP u Epsilonproteobacteria na ogół nie zawierają białek podjednostki NapC (Klotz i in., 2006; Sievert i in., 2008; Kern i Simon, 2009; Klotz i Stein, 2010; Simon i Klotz, 2013). Proponowana odwrotna funkcja kompleksu n-cM552 (CycB/NapC) jako kompleksu reduktazy azotynowej z oksydazą chinolową jest wykonalna, biorąc pod uwagę obecność obu transkryptów i brak krytycznego białkowego ligandu tyrozynowego w N. profundicola HaoA (Campbell i in., 2009), które oddzielają redukujące N-tlenki NrfA i ONR od utleniających N-tlenki kompleksów HAO (Bergmann i in., 2005; Klotz i Stein, 2008; Kern i in., 2011; Simon i Klotz, 2013).
Dane dotyczące asymilacji 15N pozwoliły ustalić, że N. profundicola włącza azot azotanowy do biomasy podczas wzrostu, gdy jest on dostarczany jako jedyne źródło azotu. Jednakże znaczna nadwyżka produkcji amonu z azotanu nad obliczonym zapotrzebowaniem na biosyntezę prowadzi nas do wniosku, że podstawową funkcją redukcji azotanu do amonu przez N. profundicola jest funkcja wydajnego oddechowego akceptora elektronów. Wniosek ten jest poparty przez zwiększone tempo wzrostu i plon w kulturach N. profundicola z azotanem jako jedynym akceptorem elektronów. Ponadto, dane dotyczące ekspresji genów zgadzają się z naszą propozycją (Campbell i in., 2009), że dzieje się to za sprawą zgodnego działania Nap, HURM (3 i cM552) oraz Har/Hcp jako nowego szlaku asymilacji N i zachowania energii: szlaku reverse-HURM. Wreszcie, dane wskazują, że N. profundicola ma zdolność do szybkiej redukcji hydroksyloaminy do amonu, co jest zgodne z propozycją wolnej hydroksyloaminy jako intermediatu w szlaku odwrotnym do HURM. Zaobserwowane wysokie tempo wychwytu hydroksyloaminy prawdopodobnie wyjaśnia, dlaczego nie jest ono konsekwentnie obserwowane podczas wzrostu kultur na azotanie. Wstępne pomiary produkcji amonu z azotanu lub azotynu w spoczywających komórkach są podobne do szybkości 3 μM min-1 (108 komórek)-1 obliczonej na podstawie hodowli, ~10-krotnie niższej niż obserwowana szybkość zużycia hydroksyloaminy. Biorąc pod uwagę te szybkości, hydroksyloamina powinna być zużywana natychmiast po wytworzeniu w hodowlach rosnących na azotanie. Ponieważ hydroksyloamina jest mutagenna, przypuszczamy, że odzwierciedla to wcześniejszą presję selekcyjną na utrzymanie niskiego poziomu hydroksyloaminy podczas wzrostu.
Próbowano znakowania biomasy za pomocą 15N-NH2OH w seryjnych hodowlach N. profundicola, ale nie zakończyło się to sukcesem (dane nie pokazane). Biorąc pod uwagę, że hydroksyloamina w hodowlach wsadowych musi być dostarczana w niskich stężeniach w stosunku do nieznakowanego azotanu, aby uniknąć toksyczności i że zdecydowana większość N z hydroksyloaminy powinna być przekształcona w amon (>90% w oparciu o Rysunek 3), wynik ten nie jest zaskakujący. Krótkotrwałe eksperymenty ze znakowaniem przy znacznie wyższych stężeniach znakowanej hydroksyloaminy jako jedynego źródła N mogą ostatecznie ustalić ten punkt.
Podsumowując, dane przedstawione tutaj są zgodne z modelem, w którym N. profundicola redukuje azotan poprzez wolne pośrednie hydroksyloaminy. Eksperymenty te nie wykluczają jeszcze całkowicie możliwości, że proponowana u N. profundicola aktywność reverse-HURM funkcjonuje jako amonotwórcza reduktaza azotynowa, która przenosi 6 elektronów bez uwalniania pośredniego N-tlenku. Niepublikowane wyniki wskazują, że kilka mutacji punktowych w genie nrfA może przekształcić reduktazę azotynu pentahemu cytochromu c w enzym nieszczelny, który uwalnia intermediaty tlenku N i nie przekształca azotynu stechiometrycznie w amon (Jörg Simon, pers. communi.). Biorąc pod uwagę ewolucyjną relację między nrfA i haoA (Bergmann i in., 2005; Simon i Klotz, 2013), moduł odwrotny-HURM u N. profundicola może naturalnie funkcjonować jako kompleks enzymatyczny produkujący amon i hydroksyloaminę, podczas gdy ekstantyczna NrfA działa „bez przecieków” jako reduktaza azotynów produkująca amon. Dalsze eksperymenty z wykorzystaniem inhibitorów Har/Hcp z azotanem lub azotynem jako substratem w całych komórkach i/lub badania biochemiczne kompleksu odwrotnego-HURM w celu określenia produktu in vitro powinny dostarczyć dodatkowych bezpośrednich testów tej hipotezy. Niezależnie od konkretnego mechanizmu, intensywna produkcja amonu znacznie przekraczająca biosyntetyczne potrzeby przez N. profundicola sugeruje, że ona i pokrewne Epsilonproteobacteria mogą pełnić ekologiczną rolę jako źródło amonu w bogatych w azotany, pozbawionych amonu, hipoksyjnych środowiskach, w których są powszechnie spotykane.
Materiały i metody
Obliczenia energii swobodnych
Energie swobodne Gibbsa (Tabela 2) obliczono ze standardowych potencjałów redoks połówek reakcji za pomocą równania Nernsta (Δ G0 = -nFE0) lub przez sumowanie energii swobodnych połówek reakcji zebranych z literatury (Thauer i in., 1977; van der Star et al., 2008). Wszystkie obliczenia przeprowadzono w standardowych warunkach (1 M wszystkich gatunków, 25°C, pH = 7).
Hodowla i analiza związków azotowych
N. profundicola szczep AmH (ATCC BAA-1463) hodowano i liczono komórki zgodnie z wcześniejszym opisem (Smith i in., 2008; Campbell i in., 2009). Amon w supernatantach hodowli oznaczano ilościowo po derywatyzacji chlorkiem dansylu i rozdzieleniu za pomocą HPLC z detekcją fluorescencyjną (Shakila i in., 2001). Azotany oznaczano ilościowo metodą HPLC z detekcją UV/Vis. Stężenia hydroksyloaminy oznaczano w filtratach hodowli (0,2 μm) zgodnie z opisem Freara i Burrella (1955).
Procedury znakowania 15N
Roztwory magazynowe 20 Atom% 15N-azotanu i 15N-amonu przygotowywano przez zmieszanie soli o stężeniu >99 Atom% i naturalnej obfitości w celu przygotowania pożywki o końcowym stężeniu 5 mM azotanu lub amonu, w zależności od potrzeb. Komórki zebrano z hodowli znakowanych 15N przez odwirowanie, a osad komórkowy przepłukano trzykrotnie wodą nanopurynkową w celu usunięcia soli. Zawiesina komórek była suszona w cynowych kapsułach, które były wysyłane do laboratorium stabilnych izotopów Uniwersytetu Kalifornijskiego w Davis, gdzie 15N Atom % był oznaczany przez spektrometrię masową stosunku izotopów. Próbki kontrolne wskazywały, że podczas przetwarzania próbek nie doszło do zanieczyszczenia izotopowego (dane nie pokazane).
Preparaty ilościowe PCR
Gene-specific primers (Tabela 3) zostały użyte do ilościowego określenia obfitości mRNA w całkowitym RNA wyekstrahowanym z N. profundicola, jak wcześniej opisano (Campbell i in., 2009).

Tabela 3. Primery i warunki do ilościowego oznaczania specyficznych genów metodą Q-RT-PCR w próbkach całkowitego RNA wyekstrahowanych z N. profundicola.
Wchłanianie hydroksyloaminy
N. profundicola hodowano na H2 i mrówczanie jako donorach elektronów oraz azotanie jako jedynym akceptorze elektronów do późnej fazy wykładniczej. Komórki były zbierane przez odwirowanie. Wszystkie transfery i płukania komórek przeprowadzono w komorze beztlenowej Coy Laboratories. Komórki ponownie zawieszano w podłożu bezazotanowym do końcowego stężenia ~109 komórek ml-1 i inkubowano w temperaturze 45°C przez 2 h w atmosferze 80% H2 + 20% CO2 przed dodaniem hydroksyloaminy (5 mM stężenie końcowe) z beztlenowo przygotowanego roztworu podstawowego. Podwielokrotności zawiesiny komórkowej były filtrowane i oznaczane dla hydroksyloaminy, jak opisano powyżej oraz dla amonu przez HPLC po derywatyzacji etoksymetylenemalonianem dietylu (Gómez-Alonso i in., 2007).
Wkład Autorów
Thomas E. Hanson, Barbara J. Campbell i Martin G. Klotz wspólnie zaprojektowali eksperymenty i zinterpretowali wyniki, Thomas E. Hanson wykonał wszystkie analizy związków azotu i przygotował próbki do spektrometrii masowej, Barbara J. Campbell wyhodowała wszystkie kultury i przeprowadziła ilościowe PCR, Katie M. Kalis przeprowadziła eksperymenty wychwytu hydroksyloaminy, Mark A. Campbell zidentyfikował NAMH_1302 jako istotny gen. Thomas E. Hanson przygotował projekt pracy, który został następnie poprawiony przez Thomasa E. Hansona, Barbarę J. Campbell i Martina G. Klotza.
Oświadczenie o konflikcie interesów
Autorzy deklarują, że badania zostały przeprowadzone przy braku jakichkolwiek komercyjnych lub finansowych relacji, które mogłyby być interpretowane jako potencjalny konflikt interesów.
Podziękowania
Autorzy chcieliby podziękować Lisie Y. Stein (Univ. Alberta-Edmonton), Jenn Macalady (Penn State Univ.), Davidowi Kirchmanowi i Mattowi Cottrellowi (Univ. Delaware) za krytyczne przeczytanie projektów manuskryptów, Jennifer Biddle za dostęp do komory beztlenowej i Kathy Coyne za dostęp do HPLC. Projekt ten był wspierany przez granty z National Science Foundation dla Barbary J. Campbell i S. Craig Cary (EF-0333203), dla Barbary J. Campbell (DEB-0640414), dla Barbary J. Campbell i Thomasa E. Hansona (MCB-0950691), oraz dla Martina G. Klotz (MCB-0948202/1202648), przez grant EPSCoR RII-2 dla University of Delaware (EPS-0814251), oraz przez fundusze motywacyjne dostarczone przez University of North Carolina dla Martina G. Klotz.
Kern, M., i Simon, J. (2009). Electron transport chains and bioenergetics of respiratory nitrogen metabolism in Wolinella succinogenes and other Epsilonproteobacteria. Biochim. Biophys. Acta 1787, 646-656. doi: 10.1016/j.bbabio.2008.12.010
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Lindsey, G. A., and Rhines, C. M. (1932). The production of hydroxylamine by the reduction of nitrates and nitrites by various pure cultures of bacteria. J. Bacteriol. 24, 489-492.
Pubmed Abstract | Pubmed Full Text
Loferer-Krossbacher, M., Klima, J., and Psenner, R. (1998). Determination of bacterial cell dry mass by transmission electron microscopy and densitometric image analysis. Appl. Environ. Microbiol. 64, 688-694.
Pubmed Abstract | Pubmed Full Text
.