Introdução
Nitrato é uma grande piscina de nitrogênio fixo no ciclo N global (Falkowski e Godfrey, 2008; Klotz e Stein, 2008). O nitrato pode ser reduzido via desnitrificação para óxido nitroso ou gás de dinitrogênio, onde é perdido dos ecossistemas terrestres e aquáticos, ou via amonificação para amônio que pode ser retido (Brandes et al., 2007; Klotz e Stein, 2008, 2010). Em ambos os caminhos, o nitrato é reduzido a nitritos. Em denitritos heterotróficos e oxidantes de amoníaco, os nitritos são reduzidos ainda mais através da formação de nitritos redutores de NO: NirS ou NirK. A redução de nitritos ao amônio é catalisada pela assimilação (NirB/NirA) ou respiratória (NrfA) de nitrito formador de nitrito redutor (Klotz e Stein, 2008, 2010). Os quimiolitrófos e metanotrófos oxidantes da amônia também podem formar NO pela oxidação da hidroxilamina, outra via potencial de perda de nitrogênio em ecossistemas terrestres e aquáticos (Campbell et al., 2011). Várias Epsilonproteobactérias que são capazes de usar nitrato para a respiração e/ou como única fonte de nitrogênio para o crescimento não codificam homólogos de qualquer tipo de nitrito redutase, sugerindo que esses organismos possuem um mecanismo não reconhecido para a redução de nitritos para amônia.
A anotação do genoma indica que não apenas Nautilia profundicola, mas vários Campylobacter spp. patogénicos entre os Epsilonproteobacteria podem utilizar o nitrato como fonte de nitrogénio para o crescimento e/ou como aceitador terminal de electrões. N. profundicola é um membro da linhagem de ramificação mais profunda das Epsilonproteobactérias (Campbell et al., 2006; Smith et al., 2008) e seus parentes só foram encontrados em ambientes de ventos hidrotermais profundos. Em contraste, as Campylobacter spp. são tipicamente associadas a hospedeiros animais e a sua presença em outros ambientes é mal definida. Enquanto os genomas de N. profundicola, Campylobacter concisus, C. curvus e C. fetus contêm genes que codificam um complexo de guanina dinucleótido de nitrato redutase (NAP) periplasmática, não há genes que codifiquem as redutases de nitrito formadoras de amônio ou NO. Além disso, um gene napC/cycB foi identificado no genoma de N. profundicola (Campbell et al., 2009), o que é incomum, pois o complexo NAP em Epsilonproteobacteria geralmente não possui uma proteína NapC (Kern e Simon, 2009; Klotz e Stein, 2010; Simon e Klotz, 2013). Com base na reconstrução metabólica informada do genoma (Campbell et al., 2009), propusemos recentemente uma nova via para a assimilação de nitratos (Figura 1) onde o nitrito é reduzido a hidroxilamina por um módulo redox hidroxilamina:ubiquinona (HURM, ou seja, uma hidroxilamina desidrogenase dependente do quinino), que denominamos via inversa ao HURM. Em N. profundicola, pensa-se que o HURM consiste num citocromo cM552 (cycB, NAMH_0559) relacionado com NapC/NrfH que medeia a transferência de electrões entre a piscina de quinonas e uma hidroxilamina oxidoredutase periplasmática (HAO, NAMH_1280). HURM, operando no sentido frontal e consistindo de hidroxilamina desidrogenase (EC:1.7.2.6; HaoA3) conectada a um vaivém de electrões de proteína citocromo c (c554 e/ou cM552 codificados por cycA e cycB, respectivamente), liga a oxidação da hidroxilamina ao pool de quinonas em bactérias aeróbias oxidantes de amónia (Klotz e Stein, 2008; Simon e Klotz, 2013). Em bactérias anaeróbias oxidantes de amônia (anammox), o módulo redox consiste em hidrazina oxidoredutase e um citocromo c ainda não identificado (Jetten et al., 2009; Simon e Klotz, 2013). Na via reversa do HURM, a hidroxilamina gerada a partir do nitrito no periplasma é transportada para o citoplasma e reduzida a amônio através de uma hidroxilamina redutase, também conhecida como proteína híbrida de cluster (Har/Hcp). Assim, as reacções propostas de aceitação de electrões da via HURM inversa são:
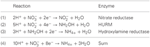
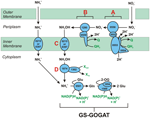
Figura 1. O caminho de HURM invertido em N. profundicola. Os passos individuais são anotados com letras maiúsculas na figura. Os números nas proteínas representadas referem-se a locus tags do genoma AmH (ou seja, 1280 = NAMH_1280 = HaoA). (A) Redução de nitrato por nitrato periplasmático respiratório redutase (NapABGH, 0556-53), (B) HURM (Hidroxilamina:Ubiquinona Redox Module) composta de uma hidroxilamina oxidoredutase em operação reversa (HaoA, 1280) funcionando como uma ocataheme nitrito redutase, que produz hidroxilamina utilizando elétrons doados a partir de um tetra-heme citocromo c da família NapC/NrfH/cM552 (CycB, 0559). (C) Transporte de hidroxilamina através de transportadores de amoníaco relacionados com AmtB. (D) Redução da hidroxilamina para amônia através de um agregado híbrido proteína/hidroxilamina redutase (Hcp/Har, 1074) utilizando a potência redutora de um Fe-S previsto contendo proteína (1302) cujo doador de elétrons (X) é atualmente desconhecido. Assimilação do amônio em biomassa ocorre via glutamina sintetase e glutamina:2-oxoglutarate aminotransferase (GS-GOGAT).
Os genomas de N. profundicola, C. concisus, C. curvus, C. fetus, e Caminibacter mediatlanticus contêm cada um genes que codificam as enzimas da via HURM inversa, enquanto os genomas de outras Epsilonproteobacteria codificam homólogos do clássico NirS/NirK formador de NO e siroheme assimilador formador de amônio NirA, ou pentaheme respiratório NrfA nitrite reductases . Hipotecamos que a via reversa-HURM melhora a sobrevivência e/ou dispersão de N. profundicola e Campylobacter spp. em ambientes hospedeiros ou não hostis com deficiência de amônia. Entretanto, para que esta hipótese seja verdadeira, a via de HURM reversa deve fornecer amônia para biossíntese e/ou facilitar a conservação de energia nestes organismos.

Tabela 1. Inventários do gene do metabolismo do nitrogênio em Epsilonproteobactérias sequenciadas por genoma.
Resultados
Energética do Crescimento em N. profundicola
N. profundicola utiliza tanto o formado quanto o hidrogênio como doadores de elétrons para o metabolismo energético e é tipicamente cultivado em polissulfeto como o aceitador de elétrons (Campbell et al, 2001; Smith et al., 2008). Cálculos sob condições padrão (1 M de todas as espécies, 25°C, pH = 7) indicam que quando o nitrato é reduzido pelas reações da via reversa do HURM, a energia livre de Gibbs (Δ G°) é substancialmente maior do que a disponível pela redução do polissulfeto (Tabela 2). Por exemplo, o Δ G° por mol de forma oxidada com nitrato é 7,4 vezes maior que o polissulfureto (-237 kJ mol-1 vs. -32 kJ mol-1). A partir deste cálculo, pode-se prever que o crescimento de N. profundicola deve ser estimulado em culturas com nitrato como o aceitador de elétrons em relação ao polissulfeto.
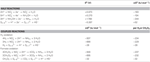
Tabela 2. Cálculo de energias livres de reacção em condições padrão (1 M todas as espécies, 25°C e pH = 7) para meias reacções de compostos de azoto e polissulfureto e reacções acopladas com formação ou hidrogénio como doador de electrões.
Crescimento de N. profundicola com nitrato
Prior caracterização e crescimento de N. profundicola utilizando meios com polissulfureto como fonte combinada de enxofre e aceitador de electrões (Campbell et al, 2001, 2009; Smith et al., 2008). Para esclarecer melhor o papel do nitrato no metabolismo energético do N. profundicola, a cepa AmH foi cultivada com sulfeto como única fonte de enxofre, de modo que o nitrato foi o único aceito de elétrons (Figura 2). O doador de elétrons era uma mistura de hidrogênio e formado em um meio sem outras fontes de enxofre ou nitrogênio (Campbell et al., 2001). As taxas de crescimento com nitrato como potencial aceitador de elétrons e fonte de nitrogênio e sulfeto (fonte de enxofre) ou polissulfeto (fonte de enxofre e potencial aceitador de elétrons) foram mais do dobro das taxas de crescimento observadas em culturas usando amônia como única fonte de nitrogênio e polissulfeto como um aceitador de elétrons e fonte de enxofre. Como esperado, culturas de controle negativo (sem aceitador de elétrons) fornecidas com amônia como fonte de nitrogênio e sulfeto como fonte de enxofre foram incapazes de crescer.
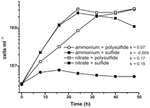
Figure 2. Crescimento de N. profundicola com aceitadores de elétrons indicados, compostos nitrogenados, e fontes de enxofre. As taxas de crescimento foram determinadas entre 8 e 24 h; k = taxa específica de crescimento constante (h-1). Cada ponto é a média de 2 ou 3 réplicas biológicas independentes.
Balanço de nitrogênio em N. profundicola Culturas
Para confirmar que o nível de amônio produzido por N. profundicola estava acima das necessidades biossintéticas, calculamos a demanda de nitrogênio biossintético da seguinte forma. Assumindo uma razão C:N de 32:6.4 para o crescimento exponencial do bacterioplâncton (Vrede et al., 2002), 288 fg de C μm-3 para o crescimento exponencial da Escherichia coli (Loferer-Krossbacher et al., 1998), e um volume celular de 0,021 μm3 calculado a partir das dimensões relatadas de N. profundicola (Smith et al., 2008), a produção de 1 × 108 células ml-1 requer ~120 ng ml-1 de N (~9 μM), um valor que não mudaria independentemente da fonte de N utilizada. N. profundicola não utilizou uma quantidade significativa da amônia fornecida durante o crescimento com polissulfeto como o aceitador de elétrons (Figura 3, círculos abertos), o que concorda com a baixa necessidade de N calculada para produzir as densidades celulares observadas. A cultura de amônia mais sulfeto não cresceu (Figura 2) e também não consumiu detectávelmente amônia (Figura 3, círculos fechados). Em contraste, a quantidade de amônia produzida por células cultivadas com 5 mM de nitrato foi 4.6 mM com polissulfeto e 5.2 mM com sulfeto (Figura 3, quadrados abertos e fechados, respectivamente). Se a via reversa do HURM na Epsilonproteobacteria serve principalmente para produzir amônia para biossíntese, nós esperaríamos baixos níveis de amônia exógena para reprimir fortemente a expressão do gene da via HURM e a atividade de redução de nitrato. Contudo, a conversão estequiométrica de nitrato em amônia em excesso da necessidade prevista de N e o consumo observado de amônia nas culturas de amônia + polissulfeto é consistente com a hipótese de que o nitrato é utilizado como um aceitador terminal de elétrons por N. profundicola e que a amonificação do nitrato não está sujeita à repressão pela amônia.
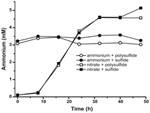
Figure 3. Concentrações de amônio medidas em culturas de N. profundicola contendo os aceitadores de elétrons indicados, compostos nitrogenados e fontes de enxofre. Cada ponto é a média de 2 ou 3 réplicas biológicas independentes.
15N-NO-3 Incorporação na Biomassa
O potencial de assimilação de nitrato foi avaliado examinando a incorporação de amônio ou nitrato marcados com 15N na biomassa de N. profundicola durante o crescimento com todas as combinações acima de doadores de elétrons e potenciais aceitadores. N. profundicola cresceu em 20 Atom% 15N de amônia rotulada com biomassa produzida com polissulfeto enriquecido a ~10 Atom% 15N após uma ronda de cultivo em lote (Figura 4), enquanto o controle não rotulado não foi enriquecido em 15N. Culturas cultivadas com sulfureto e 20 Atom% 15N de nitrato produzido a biomassa enriquecida a 16 Atom% 15N (Figura 4). A biomassa cultivada com nitrato continha aproximadamente 5 vezes mais N que a biomassa cultivada com amônia (dados não mostrados), o que pode explicar o enriquecimento mais extensivo de 15N nas amostras cultivadas com nitrato.
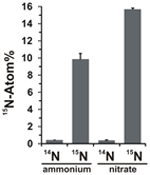
Figura 4. Absorção de nitrogênio na biomassa de N. profundicola determinada após um ciclo de cultura em lote com 5 mM dos compostos nitrogenados indicados fornecidos em abundância natural (14N) ou a 20 Atom% 15N. Os valores são médias de 3 réplicas biológicas independentes ± o desvio padrão.
Transcrição de genes-chave da via inversa
A abundância transcrita para genes que codificam as enzimas da via inversa-HURM proposta em N. profundicola foi determinada por PCR quantitativa em tempo real em culturas batch cultivadas com amônia ou nitrato. Todos os componentes da via proposta apresentaram um forte aumento na abundância transcrita em culturas cultivadas com nitrato (Figura 5). mRNAs codificando uma subunidade de nitrato redutase (napA) foram 4,6 vezes (P < 0,05, teste t heterocedástico de duas caudas) mais abundantes em células cultivadas com nitrato + sulfeto em relação às células cultivadas com amônia + polissulfeto. Transcrições de ambos os componentes do HURM, haoA e cycB/napC, foram aumentadas em 8,5 e 7,1 vezes (P < 0,05 para ambos), respectivamente. Consistente com nossa previsão de que a hidroxilamina produzida no periplasma pelo HURM seria transportada através dos principais canais facilitadores de proteína, os mRNA’s codificando ambos os homólogos do transportador de amônia/metílico amtB foram aumentados em 4,5 vezes (amtB-1, NAMH_0397, P < 0,08) e 10,3 vezes (amtB-2, NAMH_0215, P > 0,10). A hidroxilamina é um potente mutagénico que deve ser desintoxicado à medida que chega ao citoplasma. Propomos que isto seja realizado pelo Har/Hcp dependente de NADH baseado em estudos do gene hcp no cluster de genes de assimilação de nitrato de Rhodobacter capsulatus E1F1 onde é necessário para a redução e assimilação da hidroxilamina produzida por uma citoplasma assimilação de nitrato redutase (Cabello et al., 2004; Pino et al., 2006). Consistente com esta proposta, a maior mudança na abundância de transcript foi observada para o har, que aumentou 11,7 vezes, mas esta mudança não foi significativa (P > 0,10) devido à alta variabilidade nas medidas independentes da abundância de transcript do har. Originalmente, tínhamos proposto que o doador de elétrons para Har era uma NADH desidrogenase codificada por NAMH_0542 (Campbell et al., 2009). No entanto, agora acreditamos que um melhor candidato para esta função é NAMH_1302, uma proteína de cluster 4Fe-4S prevista, baseada na conservação em outras Epsilonproteobactérias (Tabela 1). Consistente com esta previsão, transcrições de NAMH_1302 foram 4,6 vezes mais abundantes em células cultivadas com nitrato em relação ao amônio, mas a mudança não foi significativa (P > 0,10).
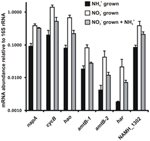
Figura 5. Níveis de mRNA em estado estável para enzimas codificadoras de genes chave do caminho inverso de HURM em N. profundicola cultivado com amônia + polissulfeto (barras pretas), nitrato + sulfeto (barras brancas), ou nitrato + polissulfeto após a adição de amônia. A abundância transcrito foi determinada por RT-PCR fluorescente e normalizada para 16S rRNA abundância determinada em cada amostra. Os valores são médias de 3 réplicas biológicas independentes ± o desvio padrão.
Para testar a potencial repressão da expressão da via pela amônia, a amônia foi adicionada a culturas de nitrato + sulfeto e a abundância de mRNA foi quantificada para os mesmos genes. Em geral, a adição de amônia diminuiu a abundância de mRNA da via reversa do HURM codificando genes de 1,2 a 3,6 vezes (Figura 5, barras cinza), embora este efeito tenha sido apenas significativo (P < 0,10) para transcrições haoA e napC/cycB que codificam as subunidades HURM. Curiosamente, a expressão de napA diminuiu o mínimo (1,2 vezes), indicando a necessidade de respiração de nitrato nestas células. Em nenhum caso, a adição de amônia reduziu os níveis de transcrição para aqueles observados em culturas cultivadas em amônia + polissulfeto.
Evidência para um intermediário de hidroxilamina
Medições diretas de hidroxilamina em filtrados de N. profundicola cultivados com nitrato encontrados baixos, mas consistentemente detectáveis níveis de hidroxilamina (5,7 ± 3,5 μM, SD, n = 3). Este baixo nível é consistente com um intermediário cujas taxas de produção e consumo são muito semelhantes.
Para determinar se a hidroxilamina poderia ser metabolizada por N. profundicola, as suspensões lavadas de células cultivadas com nitrato foram incubadas em meio de crescimento livre de nitrato com hidrogênio como único doador de elétrons. A hidroxilamina foi adicionada como potencial aceitador de elétrons e as concentrações de hidroxilamina e amônio foram seguidas ao longo do tempo. Os resultados desta experiência indicam que as células nitratadas de N. profundicola convertem rápida e completamente a hidroxilamina em amônia (Figura 6). A taxa específica de conversão de hidroxilamina neste experimento foi de 28,6 μM min-1 108 células-1. A taxa de produção de amônia a partir de nitrato calculada para crescimento exponencial na Figura 3 (8-24 h) é de ~3 μM min-1 (108 células)-1. Assim, a taxa de conversão de hidroxilamina medida é muito superior à taxa de produção de amônia observada durante o crescimento de N. profundicola com nitrato. Um acoplamento semelhante entre a produção e remoção de hidroxilamina foi relatado recentemente para o arquebactéria quimiotrofizante amônia-oxidante, Nitrosopumilus maritimus, onde a oxidação da hidroxilamina é um passo catabólico essencial (Vajrala et al., 2013).
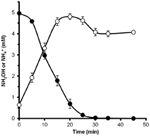
Figure 6. A hidroxilamina é rapidamente convertida em amónio por N. profundicola. As concentrações de hidroxilamina (círculos fechados) e amônia (círculos abertos) foram seguidas após a adição de 5 mM de hidroxilamina a uma suspensão de células em repouso. Os pontos são os valores médios de três réplicas ± o desvio padrão.
Discussão
Os dados aqui apresentados são consistentes com um novo caminho para a redução de nitrato em organismos que não têm a clássica formação de NO (NirS ou NirK) ou formação de nitrito de amônia (NirB/NirA) com a cepa AmH de N. profundicola como um sistema modelo útil. Uma via de HURM inversa foi proposta onde o nitrito é reduzido a hidroxilamina no periplasma e subsequentemente reduzido a amônia no citoplasma (Campbell et al., 2009). A via original foi ligeiramente modificada aqui para incluir um candidato mais provável para o doador de elétrons proximal ao Har/Hcp, um 4Fe-4S previsto contendo proteína codificada por NAMH_1302 (Figura 1). Homólogos de NAMH_1302 são conservados em C. concisus (acesso GenBank YP_001466207), C. curvus (YP_001408944), C. fetus (YP_892678), e C. mediatlanticus (ZP_01871213) que possuem todos os outros genes para a via de HURM inversa. Além disso, NAMH_1302 mostrou um aumento na abundância de transcrição em células de N. profundicola cultivadas em nitrato em relação ao amônio.
Embora a produção de hidroxilamina a partir de nitrato e nitrito tanto por via química (Rollefson e Oldershaw, 1932) como por via biológica (Lindsey e Rhines, 1932) seja há muito conhecida e um complexo de 3 foi mostrado para reduzir nitrito a hidroxilamina quando fornecido com redutor (Kostera et al, 2010), a proporção de hidroxilamina vs. produção de amónio a partir de nitrito na via inversa ao HURM ainda não é clara. A absorção de hidroxilamina extracelular pelas bactérias é conhecida; a hidroxilamina fornecida exogenamente acumula-se rapidamente no anammoxossoma de Kuenenia stuttgartiensis (Lindsay et al., 2001; Schmidt et al., 2004), um compartimento que só pode ser alcançado atravessando tanto a célula como as membranas do anammoxossoma. A captação proposta da hidroxilamina altamente mutagênica requer proteção contra lesões de DNA, o que pode explicar parcialmente o extenso complemento de sistemas de reparo de DNA identificados em N. profundicola (Campbell et al., 2009). A hidroxilamina também tem sido proposta como um intermediário da produção de amônia a partir de nitrito em plantas e algas como ferredoxina-dependente de nitrito redutase apresenta alta reatividade e especificidade para este substrato (Kuznetsova et al., 2004; Hirasawa et al, 2010).
Não parecido com a função quinona redutase do HURM em oxidantes amoniacais (Klotz e Stein, 2008; Simon e Klotz, 2013), propomos que o HURM em N. profundicola actue, semelhante ao NrfAH, como um sistema peri-plasmático de quinol oxidase que transporta electrões da piscina de quinol para reduzir o nitrito a hidroxilamina. É provável que a via inversa-HURM evoluiu de um ancestral comum do NrfAH e ONR módulo de amonificação em bactérias anammox. Hipotamos que isto ocorreu antes do HURM – incluindo um complexo trimérico de HaoA covalentemente ligado capaz de desproporção de hidroxilamina – foi usado como módulo catabólico tanto em bactérias anaeróbias como aeróbias oxidantes de amônia (Jetten et al., 2009; Klotz e Stein, 2010; Campbell et al., 2011; Kern et al., 2011; Simon e Klotz, 2013). Uma reversão semelhante da função de redução da atividade oxidante para a atividade oxidante, efetuada pela formação covalente de complexos ósseos, foi discutida para o citocromo c’-beta redutor de NO (cytS) e o citocromo P460 oxidante de NO (cytL) (Elmore et al., 2007). Os genes que codificam os módulos NrfAH e HURM reverso coexistem em apenas uma sequência de genoma bacteriano, C. fetus 82-40 (Tabela 1). Foi mostrado recentemente que a octaheme citocromo c hidroxilamina desidrogenase evoluiu como um membro de uma superfamília de proteínas multiheme citocromo c com funções nos ciclos de nitrogênio e enxofre e que o citocromo cM552 (CycB), NrfH e NapC são membros de outra superfamília citocromo c (Bergmann et al., 2005; Klotz e Stein, 2008; Kern et al., 2011; Simon e Klotz, 2013). Sabe-se também que os complexos NAP em Epsilonproteobacteria geralmente não contêm proteínas da subunidade NapC (Klotz et al., 2006; Sievert et al., 2008; Kern e Simon, 2009; Klotz e Stein, 2010; Simon e Klotz, 2013). A função inversa proposta de um complexo n-cM552 (CycB/NapC) como um complexo nitrito redutase-quinol oxidase é viável dada a presença de ambas as transcrições e a ausência da proteína ligante crítica da tirosina em N. profundicola HaoA (Campbell et al, 2009), que separam o N-óxido redutor de NrfA e ONR do complexo N-óxido oxidante HAO (Bergmann et al., 2005; Klotz e Stein, 2008; Kern et al., 2011; Simon e Klotz, 2013).
Os dados de assimilação de 15N estabeleceram que o N. profundicola incorpora nitrato-nitrogênio na biomassa durante o crescimento quando é fornecido como a única fonte de nitrogênio. No entanto, o vasto excesso de produção de amônia a partir do nitrato sobre as demandas calculadas para biossíntese nos leva a concluir que a função primária da redução de nitrato para amônia por N. profundicola é a de um eficiente aceitador de elétrons respiratórios. Esta conclusão é suportada pelo aumento das taxas de crescimento e rendimentos em culturas de N. profundicola com nitrato como o único aceitador de electrões. Além disso, os dados de expressão gênica concordam com nossa proposta (Campbell et al., 2009) de que isso ocorre através da função concertada de Nap, HURM (3 e cM552) e Har/Hcp como um novo caminho de N-assimilação e conservação de energia: o caminho inverso do HURM. Finalmente, os dados indicam que N. profundicola tem a capacidade de reduzir rapidamente a hidroxilamina ao amônio, consistente com a proposta da hidroxilamina livre como intermediário na via de HURM reversa. A alta taxa de absorção de hidroxilamina observada provavelmente explica porque não é observada consistentemente durante o crescimento de culturas com nitrato. As medições preliminares da produção de amónio a partir de nitrato ou nitrito em células em repouso são semelhantes à taxa de 3 μM min-1 (108 células)-1 calculada a partir de culturas, ~10 vezes inferior à taxa de consumo de hidroxilamina observada. Dadas estas taxas, a hidroxilamina deve ser consumida imediatamente após a produção em culturas que crescem com nitrato. Uma vez que a hidroxilamina é mutagênica, colocamos a hipótese de que isto reflete a pressão seletiva anterior para manter baixos níveis de hidroxilamina durante o crescimento.
Embora a rotulagem da biomassa tenha sido tentada com 15N-NH2OH em culturas de N. profundicola em lote, ainda não foi bem sucedida (dados não mostrados). Dado que a hidroxilamina em culturas batch deve ser fornecida em baixas concentrações em relação ao nitrato não rotulado para evitar toxicidade e que a grande maioria do N da hidroxilamina deve ser convertida em amônia (>90% baseado na Figura 3), este resultado não é surpreendente. Experimentos de rotulagem a curto prazo com concentrações muito maiores de hidroxilamina rotulada como única fonte de N pode estabelecer definitivamente este ponto.
Em conclusão, os dados aqui apresentados são consistentes com um modelo onde o N. profundicola reduz o nitrato através de um intermediário livre de hidroxilamina. Estas experiências ainda não excluem completamente a possibilidade de que a actividade de HURM invertido proposta em N. profundicola funcione como uma nitrito redutase formadora de amónio que transfere 6 electrões sem libertar um intermediário de N-óxido. Resultados não publicados indicam que algumas mutações pontuais no gene nrfA podem tornar o citocromo pentaheme c nitrito redutase numa enzima que liberta intermediários de óxido N e não consegue converter estequiometricamente o nitrito em amónio (Jörg Simon, pers. communi.). Dada a relação evolutiva entre nrfA e haoA (Bergmann et al., 2005; Simon e Klotz, 2013), o módulo reverso-HURM em N. profundicola pode funcionar naturalmente como um complexo enzimático produtor de amónio e hidroxilamina, enquanto que o NrfA existente opera “sem fugas” como uma enzima produtora de nitrito redutase de amónio. Outras experiências usando inibidores de Har/Hcp com nitrato ou nitrito como substrato em células inteiras e/ou estudos bioquímicos do complexo HURM reverso para determinar o produto in vitro devem fornecer testes directos adicionais desta hipótese. Independentemente do mecanismo específico, a produção extensiva de amônia muito acima das necessidades biossintéticas por N. profundicola sugere que ela e as Epsilonproteobactérias relacionadas podem servir um papel ecológico como fonte de amônia em ambientes ricos em nitrato, privados de amônia e hipóxicos onde eles são comumente encontrados.
Materiais e Métodos
Cálculos de Energia Livre
Energia livre de Gibbs (Tabela 2) foram calculados a partir dos potenciais redox padrão das meias-reações pela equação de Nernst (Δ G0 = -nFE0) ou pela soma das meias energias livres de reação coletadas da literatura (Thauer et al, 1977; van der Star et al., 2008). Todos os cálculos foram realizados em condições padrão (1 M todas as espécies, 25°C, pH = 7).
Cultura e Análise de Compostos de Nitrogênio
N. A cepa profunda AmH (ATCC BAA-1463) foi cultivada e as células contadas como descrito anteriormente (Smith et al., 2008; Campbell et al., 2009). O amônio em sobrenadantes de cultura foi quantificado após derivatização com cloreto de dansyl e separação por HPLC com detecção de fluorescência (Shakila et al., 2001). O nitrato foi quantificado por HPLC usando a detecção UV/Vis. As concentrações de hidroxilamina foram determinadas em filtrados de cultura (0,2 μm) como descrito por Frear e Burrell (1955).
15N Procedimentos de Rotulagem
Soluções de estoque de 20 Atom% 15N-nitrato e 15N-amônio foram preparadas misturando >99 Atom% e sais de abundância natural para preparar o meio a uma concentração final de nitrato de 5 mM ou amônio, conforme necessário. As células foram colhidas de culturas com 15N rotulado por centrifugação e o pellet de células foi lavado três vezes com água nanopurada para remover os sais. A suspensão celular foi seca em cápsulas de estanho que foram enviadas para o laboratório de isótopos estáveis da Universidade da Califórnia-Davis, onde 15N Atom % foi determinado por espectrometria de massa de razão isotópica. As amostras de controlo indicaram que não tinha ocorrido contaminação isotópica durante o processamento da amostra (dados não apresentados).
PCR quantitativa
Primers específicos do género (Tabela 3) foram utilizados para quantificar a abundância de mRNA no RNA total extraído de N. profundicola, como descrito anteriormente (Campbell et al.., 2009).

Tábua 3. Primers e condições para quantificação de Q-RT-PCR de genes específicos em amostras totais de RNA extraídos de N. profundicola.
Aumento de Hidroxilamina
N. profundicola foi cultivada em H2 e formada como doador de elétrons e nitrato como o único aceito de elétrons até a fase exponencial tardia. As células foram colhidas por centrifugação. Todas as transferências e lavagens celulares foram realizadas em uma câmara anaeróbica dos Laboratórios Coy. As células foram ressuspendidas com meio de crescimento livre de nitrato até uma concentração final de ~109 células ml-1 e incubadas a 45°C durante 2 h sob uma atmosfera de 80% H2 + 20% CO2 antes da adição de hidroxilamina (5 mM de concentração final) a partir de uma solução de reserva preparada anaerobiamente. As alíquotas da suspensão celular foram filtradas e testadas para hidroxilamina como descrito acima e para amônio por HPLC após derivatização com etoximetilenemalonato de dietila (Gómez-Alonso et al., 2007).
Author Contributions
Thomas E. Hanson, Barbara J. Campbell, e Martin G. Klotz desenharam e interpretaram em colaboração experiências, Thomas E. Hanson realizou todas as análises de compostos nitrogenados e preparou amostras para espectrometria de massa, Barbara J. Campbell cultivou todas as culturas e realizou PCR quantitativa, Katie M. Kalis realizou experiências de absorção de hidroxilamina, Mark A. Campbell identificou NAMH_1302 como um gene relevante. Thomas E. Hanson redigiu o trabalho, que foi posteriormente revisto por Thomas E. Hanson, Barbara J. Campbell e Martin G. Klotz.
Conflict of Interest Statement
Os autores declaram que a pesquisa foi conduzida na ausência de quaisquer relações comerciais ou financeiras que pudessem ser interpretadas como um potencial conflito de interesses.
Kern, M., e Simon, J. (2009). Cadeias de transporte de elétrons e bioenergética do metabolismo do nitrogênio respiratório em Wolinella succinogenes e outras Epsilonproteobacterias. Biochim. Biofísicos. Acta 1787, 646-656. doi: 10.1016/j.bbabio.2008.12.010
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Lindsey, G. A., and Rhines, C. M. (1932). A produção de hidroxilamina através da redução de nitratos e nitritos por várias culturas puras de bactérias. J. Bacteriol. 24, 489-492.
Pubmed Abstract | Pubmed Full Text
Loferer-Krossbacher, M., Klima, J., e Psenner, R. (1998). Determinação da massa seca da célula bacteriana por microscopia eletrônica de transmissão e análise densitométrica de imagens. Aplic. ambiente. Microbiol. 64, 688-694.
Pubmed Abstract | Pubmed Full Text