Introduction
Le nitrate est un pool d’azote fixe majeur dans le cycle global de l’azote (Falkowski et Godfrey, 2008 ; Klotz et Stein, 2008). Le nitrate peut être réduit par dénitrification en oxyde nitreux ou en diazote, ce qui le fait disparaître des écosystèmes terrestres et aquatiques, ou par ammonification en ammonium qui peut être retenu (Brandes et al., 2007 ; Klotz et Stein, 2008, 2010). Dans les deux voies, le nitrate est réduit en nitrite. Dans les dénitrificateurs hétérotrophes et les oxydants d’ammoniac, le nitrite est encore réduit par des nitrites réductases formant du NO : NirS ou NirK. La réduction du nitrite en ammonium est catalysée par une nitrite réductase assimilatrice (NirB/NirA) ou respiratoire (NrfA) formant de l’ammoniac (Klotz et Stein, 2008, 2010). Les chimiolithotrophes oxydant l’ammoniac et les méthanotrophes peuvent également former du NO par l’oxydation de l’hydroxylamine, une autre voie potentielle de perte d’azote dans les écosystèmes terrestres et aquatiques (Campbell et al., 2011). Plusieurs Epsilonprotéobactéries qui sont capables d’utiliser le nitrate pour la respiration et/ou comme seule source d’azote pour la croissance ne codent pas d’homologues de l’un ou l’autre type de nitrite réductase, ce qui suggère que ces organismes possèdent un mécanisme non reconnu pour la réduction du nitrite en ammonium.
L’annotation du génome indique que non seulement Nautilia profundicola, mais plusieurs Campylobacter spp. pathogènes parmi les Epsilonproteobacteria peuvent utiliser le nitrate comme source d’azote pour la croissance et/ou comme accepteur terminal d’électrons. N. profundicola est un membre de la lignée ramifiée la plus profonde des Epsilonproteobacteria (Campbell et al., 2006 ; Smith et al., 2008) et ses parents n’ont été trouvés que dans des environnements de cheminées hydrothermales en eaux profondes. En revanche, les Campylobacter spp. sont généralement associés à des hôtes animaux et leur présence dans d’autres environnements est mal définie. Alors que les génomes de N. profundicola, Campylobacter concisus, C. curvus et C. fetus contiennent des gènes codant pour un complexe nitrate-réductase périplasmique lié à la molybdoptérine guanine dinucléotide (NAP), il n’y a pas de gènes codant pour des nitrites-réductases formant de l’ammonium ou du NO. En outre, un gène napC/cycB a été identifié dans le génome de N. profundicola (Campbell et al., 2009), ce qui est inhabituel car le complexe NAP des Epsilonprotéobactéries est généralement dépourvu de la protéine NapC (Kern et Simon, 2009 ; Klotz et Stein, 2010 ; Simon et Klotz, 2013). Sur la base d’une reconstruction métabolique informée par le génome (Campbell et al., 2009), nous avons récemment proposé une nouvelle voie d’assimilation du nitrate (Figure 1) dans laquelle le nitrite est réduit en hydroxylamine par un module redox hydroxylamine:ubiquinone (HURM, c’est-à-dire une hydroxylamine déshydrogénase dépendante des quinones), que nous appelons la voie HURM inverse. Chez N. profundicola, on pense que le HURM est constitué d’un cytochrome cM552 (cycB, NAMH_0559) lié à NapC/NrfH, qui assure le transfert d’électrons entre le pool de quinone et une hydroxylamine oxydoréductase (HAO, NAMH_1280) périplasmique. L’HURM, qui opère dans le sens direct et consiste en une hydroxylamine déshydrogénase (EC:1.7.2.6 ; HaoA3) connectée à une navette électronique de la protéine cytochrome c (c554 et/ou cM552 codés par cycA et cycB, respectivement), relie l’oxydation de l’hydroxylamine au pool de quinones chez les bactéries aérobies oxydant l’ammoniac (Klotz et Stein, 2008 ; Simon et Klotz, 2013). Chez les bactéries anaérobies oxydant l’ammoniac (anammox), le module redox se compose de l’hydrazine oxydoréductase et d’un cytochrome c non encore identifié (Jetten et al., 2009 ; Simon et Klotz, 2013). Dans la voie inverse de l’HURM, l’hydroxylamine générée à partir du nitrite dans le périplasme est transportée dans le cytoplasme et réduite en ammonium via une hydroxylamine réductase, également connue sous le nom de protéine hybride (Har/Hcp). Ainsi, les réactions acceptrices d’électrons proposées de la voie de l’HURM inverse sont :
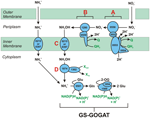
Figure 1. La voie de l’HURM inverse chez N. profundicola. Les étapes individuelles sont notées avec des lettres majuscules dans la figure. Les numéros des protéines représentées font référence aux étiquettes de locus du génome AmH (c’est-à-dire, 1280 = NAMH_1280 = HaoA). (A) Réduction du nitrate par la nitrate réductase périplasmique respiratoire (NapABGH, 0556-53), (B) HURM (Hydroxylamine :Ubiquinone Redox Module) composé d’une hydroxylamine oxydoréductase à fonctionnement inverse (HaoA, 1280) fonctionnant comme une nitrite réductase ocathème, qui produit de l’hydroxylamine en utilisant des électrons donnés par un cytochrome c tétrahème de la famille NapC/NrfH/cM552 (CycB, 0559). (C) Transport de l’hydroxylamine par des transporteurs d’ammoniac apparentés à AmtB. (D) Réduction de l’hydroxylamine en ammonium par une protéine hybride cluster/hydroxylamine réductase (Hcp/Har, 1074) utilisant le pouvoir réducteur d’une protéine prédite contenant du Fe-S (1302) dont le donneur d’électron (X) est actuellement inconnu. L’assimilation de l’ammonium dans la biomasse se produit via la glutamine synthétase et la glutamine:2-oxoglutarate aminotransférase (GS-GOGAT).
Les génomes de N. profundicola, C. concisus, C. curvus, C. fetus, et Caminibacter mediatlanticus contiennent chacun des gènes codant pour les enzymes de la voie de l’HURM inverse tandis que les génomes d’autres Epsilonproteobactéries codent pour les homologues des nitrites réductases classiques NirS/NirK formant du NO et NirA assimilateur formant de l’ammonium, ou pentahème respiratoire NrfA . Nous émettons l’hypothèse que la voie HURM inverse améliore la survie et/ou la dispersion de N. profundicola et de Campylobacter spp. dans des environnements hôtes ou non associés à des hôtes déficients en ammonium. Cependant, pour que cette hypothèse soit vraie, la voie de l’HURM inverse doit fournir de l’ammonium pour la biosynthèse et/ou faciliter la conservation de l’énergie chez ces organismes.

Tableau 1. Inventaires des gènes du métabolisme de l’azote dans les Epsilonprotéobactéries séquencées par le génome.
Résultats
Energétique de la croissance chez N. profundicola
N. profundicola utilise à la fois le formiate et l’hydrogène comme donneurs d’électrons pour le métabolisme énergétique et est typiquement cultivé sur le polysulfure comme accepteur d’électrons (Campbell et al…, 2001 ; Smith et al., 2008). Les calculs effectués dans des conditions normales (1 M de toutes les espèces, 25°C, pH = 7) indiquent que lorsque le nitrate est réduit par les réactions de la voie de l’HURM inverse, l’énergie libre de Gibbs (Δ G°) est sensiblement supérieure à celle disponible par la réduction du polysulfure (Tableau 2). Par exemple, le Δ G° par mole de formate oxydé par le nitrate est 7,4 fois plus élevé que celui du polysulfure (-237 kJ mol-1 contre -32 kJ mol-1). A partir de ce calcul, on peut prédire que la croissance de N. profundicola devrait être stimulée dans les cultures avec le nitrate comme accepteur d’électrons par rapport au polysulfure.
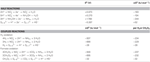
Tableau 2. Energies libres de réaction calculées dans des conditions standard (1 M toutes espèces confondues, 25°C et pH = 7) pour les demi-réactions acceptrices d’électrons des composés azotés et du polysulfure et les réactions couplées avec le formiate ou l’hydrogène comme donneur d’électrons.
Croissance de N. profundicola avec du nitrate
La caractérisation et la croissance antérieures de N. profundicola ont utilisé des milieux avec du polysulfure comme source de soufre et accepteur d’électrons combinés (Campbell et al, 2001, 2009 ; Smith et al., 2008). Pour clarifier davantage le rôle du nitrate dans le métabolisme énergétique de N. profundicola, la souche AmH a été cultivée avec du sulfure comme seule source de soufre, de sorte que le nitrate était le seul accepteur d’électrons (figure 2). Le donneur d’électrons était un mélange d’hydrogène et de formate dans un milieu sans autres sources de soufre ou d’azote (Campbell et al., 2001). Les taux de croissance avec le nitrate comme accepteur d’électrons potentiel et source d’azote et le sulfure (source de soufre) ou le polysulfure (source de soufre et accepteur d’électrons potentiel) étaient plus du double des taux de croissance observés dans les cultures utilisant l’ammonium comme seule source d’azote et le polysulfure comme accepteur d’électrons et source de soufre. Comme prévu, les cultures de contrôle négatif (sans accepteur d’électrons) fournies avec de l’ammonium comme source d’azote et du sulfure comme source de soufre n’ont pas pu croître.
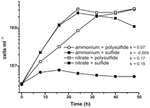
Figure 2. Croissance de N. profundicola avec les accepteurs d’électrons, les composés azotés et les sources de soufre indiqués. Les taux de croissance ont été déterminés entre 8 et 24 h ; k = constante de vitesse de croissance spécifique (h-1). Chaque point est la moyenne de 2 ou 3 répétitions biologiques indépendantes.
Equilibre azoté dans les cultures de N. profundicola
Pour confirmer que le niveau d’ammonium produit par N. profundicola était supérieur aux besoins biosynthétiques, nous avons calculé la demande d’azote biosynthétique comme suit. En supposant un rapport C:N de 32:6,4 pour le bactérioplancton à croissance exponentielle (Vrede et al., 2002), 288 fg de C μm-3 pour Escherichia coli à croissance exponentielle (Loferer-Krossbacher et al., 1998), et un volume cellulaire de 0,021 μm3 calculé à partir des dimensions rapportées de N. profundicola (Smith et al., 2008), la production de 1 × 108 cellules ml-1 nécessite ~120 ng ml-1 de N (~9 μM), un chiffre qui ne changerait pas quelle que soit la source de N utilisée. N. profundicola n’a pas utilisé une quantité importante de l’ammonium fourni pendant la croissance avec le polysulfure comme accepteur d’électrons (Figure 3, cercles ouverts), ce qui concorde avec le faible besoin en N calculé pour produire les densités cellulaires observées. La culture avec ammonium et sulfure n’a pas poussé (figure 2) et n’a pas non plus consommé d’ammonium de façon détectable (figure 3, cercles fermés). En revanche, la quantité d’ammonium produite par les cellules cultivées avec 5 mM de nitrate était de 4,6 mM avec le polysulfure et de 5,2 mM avec le sulfure (figure 3, carrés ouverts et fermés, respectivement). Si la voie HURM inverse chez les Epsilonprotéobactéries sert principalement à produire de l’ammonium pour la biosynthèse, nous nous attendrions à ce que de faibles niveaux d’ammonium exogène répriment fortement l’expression des gènes de la voie HURM et l’activité de réduction du nitrate. Cependant, la conversion stœchiométrique du nitrate en ammonium en excès du besoin prédit en N et la consommation d’ammonium observée dans les cultures d’ammonium + polysulfure est cohérente avec l’hypothèse que le nitrate est utilisé comme accepteur terminal d’électrons par N. profundicola et que l’ammonification du nitrate n’est pas soumise à la répression par l’ammonium.
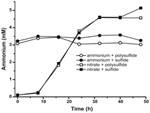
Figure 3. Concentrations d’ammonium mesurées dans des cultures de N. profundicola contenant les accepteurs d’électrons, les composés azotés et les sources de soufre indiqués. Chaque point est la moyenne de 2 ou 3 répétitions biologiques indépendantes.
Incorporation de 15N-NO-3 dans la biomasse
Le potentiel d’assimilation du nitrate a été évalué en examinant l’incorporation d’ammonium ou de nitrate marqué au 15N dans la biomasse de N. profundicola pendant la croissance avec toutes les combinaisons ci-dessus de donneurs d’électrons et d’accepteurs potentiels. N. profundicola cultivée sur de l’ammonium marqué à 20 Atom% 15N avec du polysulfure a produit une biomasse enrichie à ~10 Atom% 15N après un cycle de culture par lots (Figure 4), alors que le contrôle non marqué n’était pas enrichi en 15N. Les cultures réalisées avec du sulfure et du nitrate marqué à 20 Atom% 15N ont produit une biomasse enrichie à 16 Atom% 15N (Figure 4). La biomasse cultivée avec du nitrate contenait environ 5 fois plus de N que la biomasse cultivée avec de l’ammonium (données non présentées), ce qui peut expliquer l’enrichissement plus important en 15N dans les échantillons cultivés avec du nitrate.
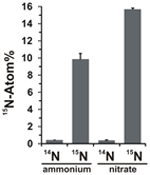
Figure 4. Absorption d’azote dans la biomasse de N. profundicola déterminée après un tour de culture en lot avec 5 mM des composés azotés indiqués fournis à l’abondance naturelle (14N) ou à 20 Atom% 15N. Les valeurs sont des moyennes de 3 répétitions biologiques indépendantes ± l’écart type.
Transcription des gènes clés de la voie
L’abondance de la transcription des gènes qui codent pour les enzymes de la voie proposée de l’HURM inverse chez N. profundicola a été déterminée par PCR quantitative en temps réel dans des cultures discontinues cultivées avec de l’ammonium ou du nitrate. Tous les composants de la voie proposée ont montré une forte augmentation de l’abondance de transcription dans les cultures de nitrate (figure 5). Les ARNm codant pour une sous-unité de nitrate réductase (napA) étaient 4,6 fois (P < 0,05, test t hétéroscédastique bilatéral) plus abondants dans les cellules cultivées de nitrate + sulfure par rapport aux cellules cultivées d’ammonium + polysulfure. Les transcrits des deux composants de l’HURM, haoA et cycB/napC, ont été augmentés de 8,5 et 7,1 fois (P < 0,05 pour les deux), respectivement. Conformément à notre prédiction selon laquelle l’hydroxylamine produite dans le périplasme par le HURM serait transportée par des canaux de protéines facilitatrices majeures, les ARNm codant pour les deux homologues du transporteur d’ammonium/méthylammonium amtB ont été multipliés par 4,5 (amtB-1, NAMH_0397, P < 0,08) et 10,3 (amtB-2, NAMH_0215, P > 0,10). L’hydroxylamine est un puissant mutagène qui doit être détoxifié dès son arrivée dans le cytoplasme. Nous proposons que cela soit accompli par Har/Hcp dépendant du NADH en nous basant sur des études du gène hcp dans le groupe de gènes d’assimilation du nitrate de Rhodobacter capsulatus E1F1 où il est requis pour la réduction et l’assimilation de l’hydroxylamine produite par une nitrate réductase assimilatrice cytoplasmique (Cabello et al., 2004 ; Pino et al., 2006). Conformément à cette proposition, le changement le plus important dans l’abondance des transcriptions a été observé pour har, qui a augmenté de 11,7 fois, mais ce changement n’était pas significatif (P > 0,10) en raison de la grande variabilité des mesures indépendantes de l’abondance des transcriptions de har. À l’origine, nous avions proposé que le donneur d’électrons de Har soit une NADH déshydrogénase codée par NAMH_0542 (Campbell et al., 2009). Cependant, nous pensons maintenant qu’un meilleur candidat pour cette fonction est NAMH_1302, une protéine prédite du cluster 4Fe-4S, basée sur la conservation dans d’autres Epsilonproteobacteria (Tableau 1). Conformément à cette prédiction, les transcrits de NAMH_1302 étaient 4,6 fois plus abondants dans les cellules cultivées au nitrate par rapport à l’ammonium, mais le changement n’était pas significatif (P > 0,10).
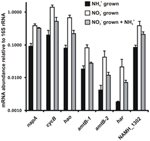
Figure 5. Niveaux d’ARNm à l’état d’équilibre pour les gènes clés codant pour les enzymes de la voie de l’HURM inverse dans N. profundicola cultivé avec de l’ammonium + polysulfure (barres noires), nitrate + sulfure (barres blanches), ou nitrate + polysulfure après l’ajout d’ammonium. L’abondance des transcrits a été déterminée par RT-PCR fluorescente et normalisée à l’abondance de l’ARNr 16S déterminée dans chaque échantillon. Les valeurs sont des moyennes de 3 répliques biologiques indépendantes ± l’écart type.
Pour tester une éventuelle répression de l’expression de la voie par l’ammonium, on a ajouté de l’ammonium à des cultures cultivées en nitrate + sulfure et on a quantifié l’abondance de l’ARNm pour les mêmes gènes. En général, l’ajout d’ammonium a diminué l’abondance de l’ARNm des gènes codant pour la voie HURM inverse de 1,2 à 3,6 fois (Figure 5, barres grises), bien que cet effet n’ait été significatif (P < 0,10) que pour les transcrits haoA et napC/cycB qui codent pour les sous-unités HURM. Fait intéressant, c’est l’expression de napA qui a le moins diminué (1,2 fois), ce qui indique la nécessité de la respiration des nitrates dans ces cellules. Dans aucun cas, l’ajout d’ammonium n’a réduit les niveaux de transcription à ceux observés dans les cultures cultivées dans l’ammonium + polysulfure.
Preuve d’un intermédiaire d’hydroxylamine
Les mesures directes d’hydroxylamine dans les filtrats des cultures de N. profundicola cultivées avec du nitrate ont trouvé des niveaux faibles, mais constamment détectables d’hydroxylamine (5,7 ± 3,5 μM, SD, n = 3). Ce faible niveau est cohérent avec un intermédiaire dont les taux de production et de consommation sont étroitement appariés.
Pour déterminer si l’hydroxylamine pouvait être métabolisée par N. profundicola, des suspensions lavées de cellules cultivées avec du nitrate ont été incubées dans un milieu de croissance sans nitrate avec de l’hydrogène comme seul donneur d’électrons. L’hydroxylamine a été ajoutée comme accepteur potentiel d’électrons et les concentrations d’hydroxylamine et d’ammonium ont été suivies dans le temps. Les résultats de cette expérience indiquent que les cellules de N. profundicola cultivées au nitrate convertissent rapidement et complètement l’hydroxylamine en ammonium (figure 6). Le taux spécifique de conversion de l’hydroxylamine dans cette expérience était de 28,6 μM min-1 108 cellules-1. Le taux de production d’ammonium à partir du nitrate calculé pour une croissance exponentielle dans la figure 3 (8-24 h) est de ~3 μM min-1 (108 cellules)-1. Ainsi, le taux de conversion de l’hydroxylamine mesuré est bien supérieur au taux de production d’ammonium observé pendant la croissance de N. profundicola avec du nitrate. Un couplage aussi étroit entre la production et l’élimination de l’hydroxylamine a récemment été signalé pour l’archée chimiolithotrophe oxydant l’ammoniac, Nitrosopumilus maritimus, où l’oxydation de l’hydroxylamine est une étape catabolique essentielle (Vajrala et al., 2013).
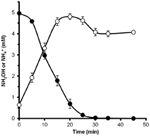
Figure 6. L’hydroxylamine est rapidement convertie en ammonium par N. profundicola. Les concentrations d’hydroxylamine (cercles fermés) et d’ammonium (cercles ouverts) ont été suivies après l’ajout de 5 mM d’hydroxylamine à une suspension cellulaire au repos. Les points sont les valeurs moyennes de trois répétitions ± l’écart-type.
Discussion
Les données présentées ici sont cohérentes avec une nouvelle voie pour la réduction du nitrate dans les organismes qui manquent de nitrite réductases classiques formant du NO (NirS ou NirK) ou formant de l’ammonium (NirB/NirA) avec la souche AmH de N. profundicola comme système modèle utile. Une voie inverse de HURM a été proposée, dans laquelle le nitrite est réduit en hydroxylamine dans le périplasme et ensuite réduit en ammonium dans le cytoplasme (Campbell et al., 2009). La voie originale a été légèrement modifiée ici pour inclure un candidat plus probable pour le donneur d’électrons proximal de Har/Hcp, une protéine contenant 4Fe-4S prédite et codée par NAMH_1302 (Figure 1). Les homologues de NAMH_1302 sont conservés dans C. concisus (accession GenBank YP_001466207), C. curvus (YP_001408944), C. fetus (YP_892678), et C. mediatlanticus (ZP_01871213) qui possèdent tous les autres gènes pour la voie inverse-HURM. En outre, NAMH_1302 a affiché une augmentation de l’abondance de la transcription dans les cellules de N. profundicola cultivées sur le nitrate par rapport à l’ammonium.
Bien que la production d’hydroxylamine à partir de nitrate et de nitrite à la fois par voie chimique (Rollefson et Oldershaw, 1932) et biologique (Lindsey et Rhines, 1932) soit connue depuis longtemps et qu’il ait été démontré qu’un complexe 3 réduit le nitrite en hydroxylamine lorsqu’il est alimenté par un réducteur (Kostera et al…, 2010), le rapport entre la production d’hydroxylamine et la production d’ammonium à partir des nitrites dans la voie de l’HURM inverse n’est pas encore clair. L’absorption de l’hydroxylamine extracellulaire par les bactéries est connue ; l’hydroxylamine fournie de manière exogène s’accumule facilement dans l’anammoxosome de Kuenenia stuttgartiensis (Lindsay et al., 2001 ; Schmidt et al., 2004), un compartiment qui ne peut être atteint qu’en traversant les membranes de la cellule et de l’anammoxosome. L’absorption proposée de l’hydroxylamine hautement mutagène nécessite une protection contre les lésions de l’ADN, ce qui peut expliquer en partie le complément important de systèmes de réparation de l’ADN identifiés chez N. profundicola (Campbell et al., 2009). L’hydroxylamine a également été proposée comme un intermédiaire de la production d’ammonium à partir du nitrite dans les plantes et les algues, car la nitrite réductase dépendante de la ferrédoxine présente une réactivité et une spécificité élevées pour ce substrat (Kuznetsova et al., 2004 ; Hirasawa et al…, 2010).
Contrairement à la fonction quinone réductase de l’HURM dans les oxydants d’ammoniac (Klotz et Stein, 2008 ; Simon et Klotz, 2013), nous proposons que l’HURM de N. profundicola agisse, comme NrfAH, comme un système quinol oxydase périplasmique qui fait la navette entre les électrons du pool de quinol pour réduire le nitrite en hydroxylamine. Il est probable que la voie inverse de l’HURM a évolué à partir d’un ancêtre commun du module d’ammonification NrfAH et ONR dans les bactéries anammox. Nous supposons que cette évolution s’est produite avant que la voie HURM – y compris un complexe trimère HaoA lié de manière covalente et capable de disproportionner l’hydroxylamine – ne soit utilisée comme module catabolique dans les bactéries anaérobies et aérobies oxydant l’ammoniac (Jetten et al., 2009 ; Klotz et Stein, 2010 ; Campbell et al., 2011 ; Kern et al., 2011 ; Simon et Klotz, 2013). Une inversion similaire de la fonction, de l’activité réductrice à l’activité oxydante, effectuée par la formation de complexes dirigés par des liaisons covalentes, a été discutée pour le cytochrome c’-beta (cytS) réducteur de NO et le cytochrome P460 (cytL) oxydant de NO (Elmore et al., 2007). Les gènes codant pour les modules NrfAH et reverse-HURM coexistent dans une seule séquence génomique bactérienne, C. fetus 82-40 (tableau 1). Il a été démontré récemment que l’hydroxylamine déshydrogénase du cytochrome c octahème a évolué en tant que membre d’une superfamille de protéines de cytochrome c multihème ayant des fonctions dans les cycles de l’azote et du soufre et que le cytochrome cM552 (CycB), NrfH, et NapC sont des membres d’une autre superfamille de cytochrome c (Bergmann et al., 2005 ; Klotz et Stein, 2008 ; Kern et al., 2011 ; Simon et Klotz, 2013). On sait également que les complexes NAP des Epsilonprotéobactéries ne contiennent généralement pas de protéines de la sous-unité NapC (Klotz et al., 2006 ; Sievert et al., 2008 ; Kern et Simon, 2009 ; Klotz et Stein, 2010 ; Simon et Klotz, 2013). La fonction inverse proposée d’un complexe n-cM552 (CycB/NapC) en tant que complexe nitrite réductase-quinol oxydase est réalisable étant donné la présence des deux transcrits et l’absence du ligand critique de la protéine tyrosine dans N. profundicola HaoA (Campbell et al., 2009), qui séparent les complexes NrfA et ONR réducteurs de N-oxyde des complexes HAO oxydants de N-oxyde (Bergmann et al., 2005 ; Klotz et Stein, 2008 ; Kern et al., 2011 ; Simon et Klotz, 2013).
Les données d’assimilation de 15N ont établi que N. profundicola incorpore l’azote des nitrates dans la biomasse pendant la croissance lorsqu’il est fourni comme seule source d’azote. Cependant, le vaste excès de production d’ammonium à partir du nitrate par rapport aux demandes calculées pour la biosynthèse nous amène à conclure que la fonction principale de la réduction du nitrate en ammonium par N. profundicola est celle d’un accepteur d’électrons respiratoire efficace. Cette conclusion est soutenue par l’augmentation des taux de croissance et des rendements dans les cultures de N. profundicola avec le nitrate comme seul accepteur d’électrons. En outre, les données sur l’expression des gènes sont en accord avec notre proposition (Campbell et al., 2009) que cela se produit via la fonction concertée de Nap, HURM (3 et cM552) et Har/Hcp comme une nouvelle voie d’assimilation de l’azote et de conservation de l’énergie : la voie inverse-HURM. Enfin, les données indiquent que N. profundicola a la capacité de réduire rapidement l’hydroxylamine en ammonium, ce qui est cohérent avec la proposition de l’hydroxylamine libre comme intermédiaire dans la voie HURM inverse. Le taux élevé d’absorption d’hydroxylamine observé explique probablement pourquoi il n’est pas observé de façon constante pendant la croissance des cultures sur nitrate. Les mesures préliminaires de la production d’ammonium à partir de nitrate ou de nitrite dans les cellules au repos sont similaires au taux de 3 μM min-1 (108 cellules)-1 calculé à partir des cultures, soit ~10 fois moins que le taux de consommation d’hydroxylamine observé. Compte tenu de ces taux, l’hydroxylamine devrait être consommée immédiatement après sa production dans les cultures se développant sur nitrate. Comme l’hydroxylamine est mutagène, nous supposons que cela reflète une pression sélective antérieure pour maintenir de faibles niveaux d’hydroxylamine pendant la croissance.
Bien que le marquage de la biomasse ait été tenté avec 15N-NH2OH dans des cultures de N. profundicola en lot, il n’a pas encore été réussi (données non présentées). Étant donné que l’hydroxylamine dans les cultures discontinues doit être fournie à de faibles concentrations par rapport au nitrate non marqué pour éviter la toxicité et que la grande majorité de l’azote provenant de l’hydroxylamine doit être convertie en ammonium (>90 % d’après la figure 3), ce résultat n’est pas surprenant. Des expériences de marquage à court terme avec des concentrations beaucoup plus élevées d’hydroxylamine marquée comme seule source de N peuvent établir définitivement ce point.
En conclusion, les données présentées ici sont cohérentes avec un modèle où N. profundicola réduit le nitrate via un intermédiaire hydroxylamine libre. Ces expériences n’excluent pas encore complètement la possibilité que l’activité HURM inverse proposée chez N. profundicola fonctionne comme une nitrite réductase formant de l’ammonium qui transfère 6 électrons sans libérer un intermédiaire N-oxyde. Des résultats non publiés indiquent que quelques mutations ponctuelles dans le gène nrfA peuvent transformer la nitrite réductase du cytochrome c pentahème en une enzyme fuyante qui libère des intermédiaires N-oxyde et ne parvient pas à convertir stoechiométriquement le nitrite en ammonium (Jörg Simon, communication personnelle). Compte tenu de la relation évolutive entre nrfA et haoA (Bergmann et al., 2005 ; Simon et Klotz, 2013), le module inverse HURM de N. profundicola pourrait naturellement fonctionner comme un complexe enzymatique produisant de l’ammonium et de l’hydroxylamine, alors que la NrfA existante fonctionne » sans fuite » comme une nitrite réductase produisant de l’ammonium. Des expériences supplémentaires utilisant des inhibiteurs de Har/Hcp avec du nitrate ou du nitrite comme substrat dans des cellules entières et/ou des études biochimiques du complexe inverse de Har/Hcp pour déterminer le produit in vitro devraient fournir des tests directs supplémentaires de cette hypothèse. Indépendamment du mécanisme spécifique, la production extensive d’ammonium bien au-delà des besoins biosynthétiques par N. profundicola suggère qu’elle et les Epsilonprotéobactéries apparentées peuvent jouer un rôle écologique en tant que source d’ammonium dans les environnements hypoxiques riches en nitrate et dépourvus d’ammonium où elles se trouvent couramment.
Matériaux et méthodes
Calculs de l’énergie libre
Les énergies libres de Gibbs (tableau 2) ont été calculées à partir des potentiels redox standard des demi-réactions par l’équation de Nernst (Δ G0 = -nFE0) ou par la sommation des énergies libres des demi-réactions recueillies dans la littérature (Thauer et al, 1977 ; van der Star et al., 2008). Tous les calculs ont été effectués dans des conditions standard (1 M toutes espèces confondues, 25°C, pH = 7).
Culture et analyse des composés azotés
La souche AmH de N. profundicola (ATCC BAA-1463) a été cultivée et les cellules comptées comme décrit précédemment (Smith et al., 2008 ; Campbell et al., 2009). L’ammonium dans les surnageants de culture a été quantifié après dérivation avec du chlorure de dansyl et séparation par HPLC avec détection de fluorescence (Shakila et al., 2001). Le nitrate a été quantifié par HPLC avec détection UV/Vis. Les concentrations d’hydroxylamine ont été déterminées dans les filtrats de culture (0,2 μm) comme décrit par Frear et Burrell (1955).
Procédures de marquage 15N
Des solutions mères de 20 Atom% de 15N-nitrate et de 15N-ammonium ont été préparées en mélangeant >99 Atom% et des sels d’abondance naturelle pour préparer le milieu à une concentration finale de 5 mM de nitrate ou d’ammonium selon les besoins. Les cellules ont été récoltées à partir de cultures marquées au 15N par centrifugation et le culot cellulaire a été lavé trois fois avec de l’eau nanopure pour éliminer les sels. La suspension cellulaire a été séchée dans des capsules d’étain qui ont été envoyées au laboratoire d’isotopes stables de l’Université de Californie-Davis où le % d’atomes de 15N a été déterminé par spectrométrie de masse du rapport isotopique. Les échantillons témoins ont indiqué qu’aucune contamination isotopique n’avait eu lieu pendant le traitement des échantillons (données non présentées).
PCR quantitative
Les amorces spécifiques aux gènes (tableau 3) ont été utilisées pour quantifier l’abondance de l’ARNm dans l’ARN total extrait de N. profundicola, comme décrit précédemment (Campbell et al, 2009).

Tableau 3. Amorces et conditions pour la quantification par Q-RT-PCR de gènes spécifiques dans des échantillons d’ARN total extraits de N. profundicola.
Prise d’hydroxylamine
N. profundicola a été cultivé sur H2 et formate comme donneurs d’électrons et nitrate comme seul accepteur d’électrons jusqu’à la phase exponentielle tardive. Les cellules ont été récoltées par centrifugation. Tous les transferts et lavages de cellules ont été effectués dans une chambre anaérobie de Coy Laboratories. Les cellules ont été remises en suspension dans un milieu de croissance sans nitrate jusqu’à une concentration finale de ~109 cellules ml-1 et incubées à 45°C pendant 2 heures sous une atmosphère de 80% H2 + 20% CO2 avant l’addition d’hydroxylamine (concentration finale de 5 mM) à partir d’une solution mère préparée en anaérobiose. Des aliquotes de la suspension cellulaire ont été filtrés et dosés pour l’hydroxylamine comme décrit ci-dessus et pour l’ammonium par HPLC après dérivation avec de l’éthoxyméthylenemalonate de diéthyle (Gómez-Alonso et al…, 2007).
Contributions des auteurs
Thomas E. Hanson, Barbara J. Campbell et Martin G. Klotz ont conçu les expériences en collaboration et interprété les résultats, Thomas E. Hanson a effectué toutes les analyses des composés azotés et préparé les échantillons pour la spectrométrie de masse, Barbara J. Campbell a cultivé toutes les cultures et effectué la PCR quantitative, Katie M. Kalis a effectué les expériences d’absorption de l’hydroxylamine, Mark A. Campbell a identifié NAMH_1302 comme un gène pertinent. Thomas E. Hanson a rédigé l’article, qui a ensuite été révisé par Thomas E. Hanson, Barbara J. Campbell et Martin G. Klotz.
Déclaration de conflit d’intérêts
Les auteurs déclarent que la recherche a été menée en l’absence de toute relation commerciale ou financière qui pourrait être interprétée comme un conflit d’intérêts potentiel.
Remerciements
Les auteurs tiennent à remercier Lisa Y. Stein (Univ. Alberta-Edmonton), Jenn Macalady (Penn State Univ.), David Kirchman et Matt Cottrell (Univ. Delaware) pour leurs lectures critiques des ébauches de manuscrits, Jennifer Biddle pour l’accès à une chambre anaérobie et Kathy Coyne pour l’accès à un HPLC. Ce projet a été soutenu par des subventions de la National Science Foundation à Barbara J. Campbell et S. Craig Cary (EF-0333203), à Barbara J. Campbell (DEB-0640414), à Barbara J. Campbell et Thomas E. Hanson (MCB-0950691), et à Martin G. Klotz (MCB-0948202/1202648), par une subvention EPSCoR RII-2 à l’Université du Delaware (EPS-0814251), et par des fonds d’incitation fournis par l’Université de Caroline du Nord à Martin G. Klotz.
Kern, M., et Simon, J. (2009). Chaînes de transport d’électrons et bioénergétique du métabolisme respiratoire de l’azote dans Wolinella succinogenes et autres Epsilonproteobacteria. Biochim. Biophys. Acta 1787, 646-656. doi : 10.1016/j.bbabio.2008.12.010
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Lindsey, G. A., et Rhines, C. M. (1932). La production d’hydroxylamine par la réduction des nitrates et des nitrites par diverses cultures pures de bactéries. J. Bacteriol. 24, 489-492.
Pubmed Abstract | Pubmed Full Text
Loferer-Krossbacher, M., Klima, J., et Psenner, R. (1998). Détermination de la masse sèche des cellules bactériennes par microscopie électronique à transmission et analyse d’image densitométrique. Appl. Environ. Microbiol. 64, 688-694.
Pubmed Abstract | Pubmed Full Text
.