Introduction
Nitrat ist ein wichtiger fixierter Stickstoffpool im globalen N-Kreislauf (Falkowski und Godfrey, 2008; Klotz und Stein, 2008). Nitrat kann durch Denitrifikation zu Distickstoffoxid oder Distickstoffgas reduziert werden, wodurch es aus terrestrischen und aquatischen Ökosystemen verloren geht, oder durch Ammonifikation zu Ammonium, das gespeichert werden kann (Brandes et al., 2007; Klotz und Stein, 2008, 2010). Bei beiden Wegen wird Nitrat zu Nitrit reduziert. In heterotrophen Denitrifikanten und Ammoniakoxidatoren wird Nitrit durch NO-bildende Nitritreduktasen weiter reduziert: NirS oder NirK. Die Reduktion von Nitrit zu Ammonium wird durch assimilatorische (NirB/NirA) oder respiratorische (NrfA) ammoniakbildende Nitritreduktase katalysiert (Klotz und Stein, 2008, 2010). Ammoniak-oxidierende Chemolithotrophe und Methanotrophe können auch NO durch die Oxidation von Hydroxylamin bilden, ein weiterer möglicher Weg des Stickstoffverlustes in terrestrischen und aquatischen Ökosystemen (Campbell et al., 2011). Mehrere Epsilonproteobakterien, die in der Lage sind, Nitrat für die Atmung und/oder als einzige Stickstoffquelle für das Wachstum zu nutzen, kodieren keine Homologe für einen der beiden Nitritreduktase-Typen, was darauf hindeutet, dass diese Organismen einen unerkannten Mechanismus zur Nitritreduktion zu Ammonium besitzen.
Genom-Annotation zeigt, dass nicht nur Nautilia profundicola, sondern auch mehrere pathogene Campylobacter spp. unter den Epsilonproteobakterien Nitrat als Stickstoffquelle für das Wachstum und/oder als terminalen Elektronenakzeptor nutzen können. N. profundicola ist ein Mitglied der am tiefsten verzweigten Linie der Epsilonproteobakterien (Campbell et al., 2006; Smith et al., 2008), und seine Verwandten wurden bisher nur in hydrothermalen Schlotumgebungen der Tiefsee gefunden. Im Gegensatz dazu sind Campylobacter spp. typischerweise mit tierischen Wirten assoziiert, und ihr Vorkommen in anderen Umgebungen ist unklar. Während die Genome von N. profundicola, Campylobacter concisus, C. curvus und C. fetus Gene enthalten, die für einen periplasmatischen Molybdopterin-Guanin-Dinukleotid-gebundenen Nitratreduktase-Komplex (NAP) kodieren, gibt es keine Gene, die für Ammonium- oder NO-bildende Nitritreduktasen kodieren. Darüber hinaus wurde im Genom von N. profundicola ein napC/cycB-Gen identifiziert (Campbell et al., 2009), was ungewöhnlich ist, da dem NAP-Komplex in Epsilonproteobakterien normalerweise ein NapC-Protein fehlt (Kern und Simon, 2009; Klotz und Stein, 2010; Simon und Klotz, 2013). Auf der Grundlage einer genominformierten metabolischen Rekonstruktion (Campbell et al., 2009) haben wir kürzlich einen neuen Weg für die Nitratassimilation vorgeschlagen (Abbildung 1), bei dem Nitrit durch ein Hydroxylamin:Ubichinon-Redoxmodul (HURM, d. h. eine chinonabhängige Hydroxylamin-Dehydrogenase) zu Hydroxylamin reduziert wird, was wir als Reverse-HURM-Weg bezeichnen. In N. profundicola besteht das HURM vermutlich aus einem NapC/NrfH-verwandten Cytochrom cM552 (cycB, NAMH_0559), das den Elektronentransfer zwischen dem Chinonpool und einer periplasmatischen Hydroxylamin-Oxidoreduktase (HAO, NAMH_1280) vermittelt. HURM, das in Vorwärtsrichtung arbeitet und aus Hydroxylamin-Dehydrogenase (EC:1.7.2.6; HaoA3) besteht, die mit einem Cytochrom-c-Protein-Elektronen-Shuttle (c554 und/oder cM552, kodiert von cycA bzw. cycB) verbunden ist, verbindet die Oxidation von Hydroxylamin mit dem Chinon-Pool in aeroben Ammoniak-oxidierenden Bakterien (Klotz und Stein, 2008; Simon und Klotz, 2013). In anaeroben ammoniakoxidierenden Bakterien (Anammox) besteht das Redoxmodul aus Hydrazin-Oxidoreduktase und einem noch nicht identifizierten Cytochrom c (Jetten et al., 2009; Simon und Klotz, 2013). Im umgekehrten HURM-Weg wird aus Nitrit im Periplasma erzeugtes Hydroxylamin ins Zytoplasma transportiert und über eine Hydroxylamin-Reduktase, auch bekannt als Hybrid-Cluster-Protein (Har/Hcp), zu Ammonium reduziert. Die vorgeschlagenen elektronenaufnehmenden Reaktionen des Reverse-HURM-Wegs sind also:
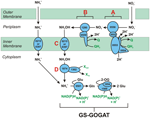
Abbildung 1. Der Reverse-HURM-Weg in N. profundicola. Die einzelnen Schritte sind in der Abbildung mit Großbuchstaben gekennzeichnet. Die Zahlen in den abgebildeten Proteinen beziehen sich auf Locus-Tags aus dem AmH-Genom (d. h. 1280 = NAMH_1280 = HaoA). (A) Nitratreduktion durch respiratorische periplasmatische Nitratreduktase (NapABGH, 0556-53), (B) HURM (Hydroxylamine:Ubichinon-Redox-Modul), bestehend aus einer umgekehrt arbeitenden Hydroxylamin-Oxidoreduktase (HaoA, 1280), die als Ocataheme-Nitrit-Reduktase fungiert und Hydroxylamin unter Verwendung von Elektronen produziert, die von einem Tetra-Hem-Cytochrom c der NapC/NrfH/cM552-Familie (CycB, 0559) abgegeben werden. (C) Transport von Hydroxylamin über Ammoniaktransporter, die mit AmtB verwandt sind. (D) Reduktion von Hydroxylamin zu Ammonium durch ein hybrides Clusterprotein/Hydroxylamin-Reduktase (Hcp/Har, 1074), das die reduzierende Kraft eines Fe-S-haltigen Proteins (1302) nutzt, dessen Elektronendonor (X) derzeit unbekannt ist. Die Assimilation von Ammonium in Biomasse erfolgt über Glutaminsynthetase und Glutamin:2-Oxoglutarat-Aminotransferase (GS-GOGAT).
Die Genome von N. profundicola, C. concisus, C. curvus, C. fetus und Caminibacter mediatlanticus enthalten jeweils Gene, die für die Enzyme des reversen HURM-Wegs kodieren, während die Genome anderer Epsilonproteobakterien Homologe der klassischen NO-bildenden NirS/NirK und des Ammonium-bildenden assimilatorischen Sirohems NirA oder der respiratorischen Pentahems NrfA-Nitritreduktasen kodieren. Wir stellen die Hypothese auf, dass der umgekehrte HURM-Weg das Überleben und/oder die Ausbreitung von N. profundicola und Campylobacter spp. in Wirtsumgebungen mit Ammoniummangel oder ohne Wirtsassoziation verbessert. Damit diese Hypothese jedoch zutrifft, muss der Reverse-HURM-Weg Ammonium für die Biosynthese bereitstellen und/oder die Energieerhaltung in diesen Organismen erleichtern.

Tabelle 1. Stickstoff-Stoffwechsel-Geninventare in genomsequenzierten Epsilonproteobakterien.
Ergebnisse
Energetik des Wachstums in N. profundicola
N. profundicola nutzt sowohl Formiat als auch Wasserstoff als Elektronendonatoren für den Energiestoffwechsel und wächst typischerweise auf Polysulfid als Elektronenakzeptor (Campbell et al., 2001; Smith et al., 2008). Berechnungen unter Standardbedingungen (1 M aller Spezies, 25 °C, pH = 7) deuten darauf hin, dass die freie Gibbs-Energie (Δ G°) bei der Reduktion von Nitrat durch die Reaktionen des umgekehrten HURM-Wegs wesentlich größer ist als bei der Reduktion von Polysulfid (Tabelle 2). Beispielsweise ist die Δ G° pro Mol mit Nitrat oxidiertem Formiat um das 7,4-fache höher als bei Polysulfid (-237 kJ mol-1 vs. -32 kJ mol-1). Aus dieser Berechnung kann man vorhersagen, dass das Wachstum von N. profundicola in Kulturen mit Nitrat als Elektronenakzeptor im Vergleich zu Polysulfid stimuliert werden sollte.
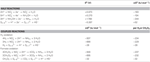
Tabelle 2. Berechnete freie Reaktionsenergien unter Standardbedingungen (1 M alle Spezies, 25°C und pH = 7) für elektronenakzeptierende Halbreaktionen von Stickstoffverbindungen und Polysulfid und gekoppelte Reaktionen mit Formiat oder Wasserstoff als Elektronendonor.
Wachstum von N. profundicola mit Nitrat
Frühere Charakterisierungen und Wachstum von N. profundicola nutzten Medien mit Polysulfid als kombinierte Schwefelquelle und Elektronenakzeptor (Campbell et al, 2001, 2009; Smith et al., 2008). Um die Rolle von Nitrat im Energiestoffwechsel von N. profundicola weiter zu klären, wurde der Stamm AmH mit Sulfid als einziger Schwefelquelle gezüchtet, so dass Nitrat der einzige Elektronenakzeptor war (Abbildung 2). Der Elektronendonator war eine Mischung aus Wasserstoff und Formiat in einem Medium ohne andere Schwefel- oder Stickstoffquellen (Campbell et al., 2001). Die Wachstumsraten mit Nitrat als potenziellem Elektronenakzeptor und Stickstoffquelle und Sulfid (Schwefelquelle) oder Polysulfid (Schwefelquelle und potenzieller Elektronenakzeptor) waren mehr als doppelt so hoch wie die Wachstumsraten, die in Kulturen mit Ammonium als einziger Stickstoffquelle und Polysulfid als Elektronenakzeptor und Schwefelquelle beobachtet wurden. Wie erwartet konnten die Kulturen der Negativkontrolle (ohne Elektronenakzeptor), die mit Ammonium als Stickstoffquelle und Sulfid als Schwefelquelle versorgt wurden, nicht wachsen.
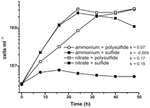
Abbildung 2. Wachstum von N. profundicola mit den angegebenen Elektronenakzeptoren, Stickstoffverbindungen und Schwefelquellen. Die Wachstumsraten wurden zwischen 8 und 24 Stunden bestimmt; k = spezifische Wachstumsratenkonstante (h-1). Jeder Punkt ist der Durchschnitt von 2 oder 3 unabhängigen biologischen Replikaten.
Stickstoffbilanz in N. profundicola-Kulturen
Um zu bestätigen, dass die von N. profundicola produzierte Ammoniummenge den biosynthetischen Bedarf übersteigt, berechneten wir den biosynthetischen Stickstoffbedarf wie folgt. Unter der Annahme eines C:N-Verhältnisses von 32:6,4 für exponentiell wachsendes Bakterioplankton (Vrede et al., 2002), 288 fg C μm-3 für exponentiell wachsende Escherichia coli (Loferer-Krossbacher et al., 1998) und einem Zellvolumen von 0,021 μm3, berechnet aus den angegebenen Abmessungen von N. profundicola (Smith et al., 2008), erfordert die Produktion von 1 × 108 Zellen ml-1 ~120 ng ml-1 N (~9 μM), eine Zahl, die sich unabhängig von der verwendeten N-Quelle nicht ändern würde. N. profundicola nutzte keine nennenswerte Menge des Ammoniums, das während des Wachstums mit Polysulfid als Elektronenakzeptor bereitgestellt wurde (Abbildung 3, offene Kreise), was mit dem niedrigen N-Bedarf übereinstimmt, der zur Erzeugung der beobachteten Zelldichten berechnet wurde. Die Ammonium-plus-Sulfid-Kultur wuchs nicht (Abbildung 2) und verbrauchte auch nicht nachweislich Ammonium (Abbildung 3, geschlossene Kreise). Im Gegensatz dazu betrug die von den mit 5 mM Nitrat gewachsenen Zellen produzierte Ammoniummenge 4,6 mM mit Polysulfid und 5,2 mM mit Sulfid (Abbildung 3, offene bzw. geschlossene Quadrate). Wenn der umgekehrte HURM-Weg in den Epsilonproteobakterien in erster Linie dazu dient, Ammonium für die Biosynthese zu produzieren, würden wir erwarten, dass geringe Mengen an exogenem Ammonium die Genexpression des HURM-Wegs und die Nitratreduktionsaktivität stark unterdrücken. Die stöchiometrische Umwandlung von Nitrat in Ammonium, die den vorhergesagten N-Bedarf übersteigt, und der beobachtete Ammoniumverbrauch in den Ammonium+Polysulfid-Kulturen stimmen jedoch mit der Hypothese überein, dass Nitrat von N. profundicola als terminaler Elektronenakzeptor genutzt wird und dass die Nitrat-Ammonifikation nicht durch Ammonium unterdrückt wird.
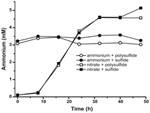
Abbildung 3. Ammoniumkonzentrationen, gemessen in Kulturen von N. profundicola, die die angegebenen Elektronenakzeptoren, Stickstoffverbindungen und Schwefelquellen enthalten. Jeder Punkt ist der Durchschnitt von 2 oder 3 unabhängigen biologischen Wiederholungen.
Einbau von 15N-NO-3 in die Biomasse
Das Potenzial für die Nitratassimilation wurde durch Untersuchung des Einbaus von 15N-markiertem Ammonium oder Nitrat in die Biomasse von N. profundicola während des Wachstums mit allen oben genannten Kombinationen von Elektronendonatoren und potenziellen Akzeptoren bewertet. N. profundicola, das mit 20 Atom-% 15N-markiertem Ammonium und Polysulfid wuchs, produzierte nach einer Runde Batch-Kultivierung Biomasse, die auf ~10 Atom-% 15N angereichert war (Abbildung 4), während die unmarkierte Kontrolle nicht mit 15N angereichert war. Kulturen, die mit Sulfid und 20 Atom-% 15N-markiertem Nitrat gezüchtet wurden, produzierten Biomasse, die auf 16 Atom-% 15N angereichert war (Abbildung 4). Die mit Nitrat gewachsene Biomasse enthielt etwa 5-mal mehr N als die mit Ammonium gewachsene Biomasse (Daten nicht gezeigt), was die stärkere Anreicherung von 15N in den mit Nitrat gewachsenen Proben erklären könnte.
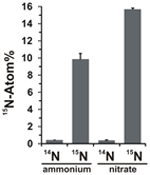
Abbildung 4. Stickstoffaufnahme in die Biomasse von N. profundicola, bestimmt nach einer Runde Batch-Kultur mit 5 mM der angegebenen Stickstoffverbindungen, die in natürlicher Häufigkeit (14N) oder mit 20 Atom% 15N zugeführt wurden. Die Werte sind Durchschnittswerte von 3 unabhängigen biologischen Wiederholungen ± Standardabweichung.
Transkription von Schlüsselgenen des Stoffwechselwegs
Die Transkriptionshäufigkeit von Genen, die für Enzyme des vorgeschlagenen Reverse-HURM-Stoffwechselwegs in N. profundicola kodieren, wurde mittels quantitativer Echtzeit-PCR in Batch-Kulturen bestimmt, die mit Ammonium oder Nitrat gezüchtet wurden. Alle Komponenten des vorgeschlagenen Weges zeigten einen starken Anstieg der Transkriptabundanz in mit Nitrat gezüchteten Kulturen (Abbildung 5). mRNAs, die für eine Nitratreduktase-Untereinheit (napA) kodieren, waren in mit Nitrat und Sulfid gezüchteten Zellen im Vergleich zu mit Ammonium und Polysulfid gezüchteten Zellen 4,6-mal (P < 0,05, zweiseitiger heteroskedastischer t-Test) häufiger. Die Transkripte der beiden Komponenten des HURM, haoA und cycB/napC, waren um das 8,5- bzw. 7,1-fache erhöht (P < 0,05 für beide). Im Einklang mit unserer Vorhersage, dass Hydroxylamin, das im Periplasma durch den HURM produziert wird, durch wichtige Vermittlerproteinkanäle transportiert wird, waren die mRNAs, die für beide Homologe des Ammonium/Methylammonium-Transporters amtB kodieren, um das 4,5-fache (amtB-1, NAMH_0397, P < 0,08) und 10,3-fache (amtB-2, NAMH_0215, P > 0,10) erhöht. Hydroxylamin ist ein starkes Mutagen, das entgiftet werden muss, sobald es im Zytoplasma ankommt. Wir vermuten, dass dies durch das NADH-abhängige Har/Hcp geschieht, basierend auf Untersuchungen des hcp-Gens im Nitratassimilations-Gencluster von Rhodobacter capsulatus E1F1, wo es für die Reduktion und Assimilation von Hydroxylamin erforderlich ist, das von einer zytoplasmatischen assimilatorischen Nitratreduktase produziert wird (Cabello et al., 2004; Pino et al., 2006). In Übereinstimmung mit diesem Vorschlag wurde die größte Veränderung der Transkriptmenge für har festgestellt, die um das 11,7-fache anstieg, aber diese Veränderung war aufgrund der hohen Variabilität der unabhängigen Messungen der har-Transkriptmenge nicht signifikant (P > 0,10). Ursprünglich hatten wir vorgeschlagen, dass der Elektronendonor für Har eine NADH-Dehydrogenase ist, die von NAMH_0542 kodiert wird (Campbell et al., 2009). Jetzt glauben wir jedoch, dass NAMH_1302, ein vorhergesagtes 4Fe-4S-Cluster-Protein, aufgrund der Konservierung in anderen Epsilonproteobakterien ein besserer Kandidat für diese Funktion ist (Tabelle 1). In Übereinstimmung mit dieser Vorhersage waren die Transkripte von NAMH_1302 in mit Nitrat gezüchteten Zellen 4,6-mal häufiger als in mit Ammonium gezüchteten, aber die Veränderung war nicht signifikant (P > 0,10).
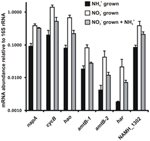
Abbildung 5. Steady-State-mRNA-Konzentrationen für Schlüsselgene, die für Enzyme des Reverse-HURM-Wegs in N. profundicola kodieren, die mit Ammonium + Polysulfid (schwarze Balken), Nitrat + Sulfid (weiße Balken) oder Nitrat + Polysulfid nach der Zugabe von Ammonium gewachsen sind. Die Transkriptionshäufigkeit wurde mittels fluoreszierender RT-PCR bestimmt und auf die in jeder Probe ermittelte 16S rRNA-Häufigkeit normiert. Die Werte sind Durchschnittswerte von 3 unabhängigen biologischen Replikaten ± Standardabweichung.
Um zu prüfen, ob Ammonium die Expression des Stoffwechselweges möglicherweise unterdrückt, wurde Ammonium zu Nitrat + Sulfid gewachsenen Kulturen hinzugefügt und die mRNA-Häufigkeit für die gleichen Gene quantifiziert. Im Allgemeinen verringerte die Ammoniumzugabe die mRNA-Häufigkeit der Gene, die für den Reverse-HURM-Stoffwechsel kodieren, um das 1,2- bis 3,6-fache (Abbildung 5, graue Balken), obwohl dieser Effekt nur für die Transkripte haoA und napC/cycB, die für die HURM-Untereinheiten kodieren, signifikant war (P < 0,10). Interessanterweise nahm die Expression von napA am wenigsten ab (1,2-fach), was auf die Notwendigkeit der Nitratatmung in diesen Zellen hinweist. In keinem Fall führte die Zugabe von Ammonium zu einer Verringerung der Transkriptmengen auf das Niveau von Kulturen, die mit Ammonium und Polysulfid gezüchtet wurden.
Beweise für ein Hydroxylamin-Zwischenprodukt
Direkte Messungen von Hydroxylamin in Filtraten von N. profundicola-Kulturen, die mit Nitrat gezüchtet wurden, ergaben geringe, aber durchweg nachweisbare Hydroxylaminmengen (5,7 ± 3,5 μM, SD, n = 3). Dieser niedrige Wert steht im Einklang mit einem Zwischenprodukt, dessen Produktions- und Verbrauchsraten eng aufeinander abgestimmt sind.
Um festzustellen, ob Hydroxylamin von N. profundicola verstoffwechselt werden kann, wurden gewaschene Suspensionen von mit Nitrat gewachsenen Zellen in nitratfreiem Wachstumsmedium mit Wasserstoff als einzigem Elektronendonor inkubiert. Hydroxylamin wurde als potenzieller Elektronenakzeptor zugegeben, und die Konzentrationen von Hydroxylamin und Ammonium wurden über die Zeit verfolgt. Die Ergebnisse dieses Versuchs zeigen, dass die mit Nitrat gewachsenen Zellen von N. profundicola Hydroxylamin schnell und vollständig in Ammonium umwandeln (Abbildung 6). Die spezifische Rate der Hydroxylaminumwandlung betrug in diesem Experiment 28,6 μM min-1 108 Zellen-1. Die für das exponentielle Wachstum in Abbildung 3 (8-24 h) berechnete Ammoniumproduktionsrate aus Nitrat beträgt ~3 μM min-1 (108 Zellen)-1. Somit liegt die gemessene Hydroxylamin-Umsetzungsrate weit über der während des Wachstums von N. profundicola mit Nitrat beobachteten Ammoniumproduktionsrate. Eine ähnlich enge Kopplung zwischen Hydroxylaminproduktion und -abbau wurde kürzlich für das chemolithotrophe ammoniakoxidierende Archaeon Nitrosopumilus maritimus berichtet, bei dem die Hydroxylaminoxidation ein wesentlicher katabolischer Schritt ist (Vajrala et al., 2013).
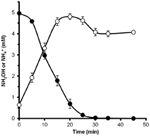
Abbildung 6. Hydroxylamin wird von N. profundicola schnell in Ammonium umgewandelt. Die Konzentrationen von Hydroxylamin (geschlossene Kreise) und Ammonium (offene Kreise) wurden nach der Zugabe von 5 mM Hydroxylamin zu einer ruhenden Zellsuspension verfolgt. Die Punkte sind die Mittelwerte aus drei Wiederholungen ± Standardabweichung.
Diskussion
Die hier vorgestellten Daten stehen im Einklang mit einem neuen Weg der Nitratreduktion in Organismen, denen klassische NO-bildende (NirS oder NirK) oder Ammonium-bildende (NirB/NirA) Nitritreduktasen fehlen, wobei der N. profundicola-Stamm AmH ein nützliches Modellsystem darstellt. Es wurde ein umgekehrter HURM-Weg vorgeschlagen, bei dem Nitrit im Periplasma zu Hydroxylamin reduziert und anschließend im Zytoplasma zu Ammonium reduziert wird (Campbell et al., 2009). Der ursprüngliche Weg wurde hier leicht modifiziert, um einen wahrscheinlicheren Kandidaten für den proximalen Elektronendonor für Har/Hcp einzubeziehen, ein 4Fe-4S-haltiges Protein, das von NAMH_1302 kodiert wird (Abbildung 1). Homologe von NAMH_1302 sind in C. concisus (GenBank-Zugang YP_001466207), C. curvus (YP_001408944), C. fetus (YP_892678) und C. mediatlanticus (ZP_01871213) konserviert, die alle anderen Gene für den Reverse-HURM-Weg besitzen. Darüber hinaus zeigte NAMH_1302 eine erhöhte Transkriptabundanz in N. profundicola-Zellen, die auf Nitrat im Vergleich zu Ammonium wuchsen.
Während die Produktion von Hydroxylamin aus Nitrat und Nitrit sowohl auf chemischem (Rollefson und Oldershaw, 1932) als auch auf biologischem Wege (Lindsey und Rhines, 1932) seit langem bekannt ist und ein 3-Komplex nachgewiesen wurde, der Nitrit zu Hydroxylamin reduziert, wenn er mit einem Reduktionsmittel versorgt wird (Kostera et al., 2010), ist das Verhältnis zwischen der Hydroxylamin- und der Ammoniumproduktion aus Nitrit im umgekehrten HURM-Weg noch nicht klar. Die Aufnahme von extrazellulärem Hydroxylamin durch Bakterien ist bekannt; exogen zugeführtes Hydroxylamin reichert sich leicht im Anammoxosom von Kuenenia stuttgartiensis an (Lindsay et al., 2001; Schmidt et al., 2004), einem Kompartiment, das nur erreicht werden kann, wenn sowohl die Zell- als auch die Anammoxosom-Membranen durchquert werden. Die vorgeschlagene Aufnahme des hochgradig mutagenen Hydroxylamins erfordert einen Schutz vor DNA-Läsionen, was teilweise die umfangreichen DNA-Reparatursysteme erklären könnte, die in N. profundicola identifiziert wurden (Campbell et al., 2009). Hydroxylamin wurde auch als Zwischenprodukt der Ammoniumproduktion aus Nitrit in Pflanzen und Algen vorgeschlagen, da die ferredoxinabhängige Nitritreduktase eine hohe Reaktivität und Spezifität für dieses Substrat aufweist (Kuznetsova et al., 2004; Hirasawa et al., 2010).
Im Gegensatz zur Chinon-Reduktase-Funktion von HURM in Ammoniak-Oxidationsmitteln (Klotz und Stein, 2008; Simon und Klotz, 2013) schlagen wir vor, dass HURM in N. profundicola, ähnlich wie NrfAH, als periplasmatisches Chinol-Oxidase-System fungiert, das Elektronen aus dem Chinol-Pool transportiert, um Nitrit zu Hydroxylamin zu reduzieren. Es ist wahrscheinlich, dass sich der Reverse-HURM-Weg aus einem gemeinsamen Vorfahren des NrfAH- und ONR-Ammonifikationsmoduls in Anammox-Bakterien entwickelt hat. Wir vermuten, dass dies geschah, bevor HURM – einschließlich eines kovalent gebundenen trimeren HaoA-Komplexes, der zur Disproportionierung von Hydroxylamin fähig ist – als kataboles Modul sowohl in anaeroben als auch in aeroben Ammoniak-oxidierenden Bakterien verwendet wurde (Jetten et al., 2009; Klotz und Stein, 2010; Campbell et al., 2011; Kern et al., 2011; Simon und Klotz, 2013). Eine ähnliche Umkehrung der Funktion von reduzierender zu oxidierender Aktivität, die durch kovalente bindungsgesteuerte Komplexbildung bewirkt wird, wurde für das NO-reduzierende Cytochrom c‘-beta (cytS) und das NO-oxidierende Cytochrom P460 (cytL) diskutiert (Elmore et al., 2007). Gene, die für die NrfAH- und Reverse-HURM-Module kodieren, existieren nur in einer einzigen bakteriellen Genomsequenz, C. fetus 82-40 (Tabelle 1). Kürzlich wurde gezeigt, dass sich die Octaheme-Cytochrom-c-Hydroxylamin-Dehydrogenase als Mitglied einer Multi-Hem-Cytochrom-c-Protein-Superfamilie mit Funktionen im Stickstoff- und Schwefelzyklus entwickelt hat und dass Cytochrom cM552 (CycB), NrfH und NapC Mitglieder einer anderen Cytochrom-c-Superfamilie sind (Bergmann et al., 2005; Klotz und Stein, 2008; Kern et al., 2011; Simon und Klotz, 2013). Es ist auch bekannt, dass NAP-Komplexe in Epsilonproteobakterien im Allgemeinen keine NapC-Untereinheiten enthalten (Klotz et al., 2006; Sievert et al., 2008; Kern und Simon, 2009; Klotz und Stein, 2010; Simon und Klotz, 2013). Die vorgeschlagene umgekehrte Funktion eines n-cM552 (CycB/NapC)-Komplexes als Nitrit-Reduktase-Chinol-Oxidase-Komplex ist angesichts des Vorhandenseins beider Transkripte und des Fehlens des kritischen Tyrosin-Proteinliganden in N. profundicola HaoA (Campbell et al., 2009), die die N-Oxid-reduzierenden NrfA- und ONR-Komplexe von den N-Oxid-oxidierenden HAO-Komplexen trennen (Bergmann et al., 2005; Klotz und Stein, 2008; Kern et al., 2011; Simon und Klotz, 2013).
Die Daten zur 15N-Assimilation belegen, dass N. profundicola während des Wachstums Nitrat-Stickstoff in die Biomasse aufnimmt, wenn es als einzige Stickstoffquelle zur Verfügung steht. Die enorme Überproduktion von Ammonium aus Nitrat gegenüber dem berechneten Bedarf für die Biosynthese lässt jedoch den Schluss zu, dass die primäre Funktion der Nitratreduktion zu Ammonium durch N. profundicola die eines effizienten Atmungselektronenakzeptors ist. Diese Schlussfolgerung wird durch die erhöhten Wachstumsraten und Erträge in N. profundicola-Kulturen mit Nitrat als einzigem Elektronenakzeptor bestätigt. Darüber hinaus stimmen die Genexpressionsdaten mit unserem Vorschlag (Campbell et al., 2009) überein, dass dies über die konzertierte Funktion von Nap, HURM (3 und cM552) und Har/Hcp als neuartiger N-Assimilations- und Energieerhaltungsweg geschieht: der Reverse-HURM-Weg. Schließlich deuten die Daten darauf hin, dass N. profundicola in der Lage ist, Hydroxylamin schnell zu Ammonium zu reduzieren, was mit dem Vorschlag übereinstimmt, freies Hydroxylamin als Zwischenprodukt im Reverse-HURM-Weg zu verwenden. Die hohe Aufnahmerate von Hydroxylamin erklärt wahrscheinlich, warum sie beim Wachstum von Kulturen auf Nitrat nicht durchgängig beobachtet wird. Vorläufige Messungen der Ammoniumproduktion aus Nitrat oder Nitrit in ruhenden Zellen ähneln der aus Kulturen berechneten Rate von 3 μM min-1 (108 Zellen)-1, die etwa 10-mal niedriger ist als die beobachtete Hydroxylaminverbrauchsrate. Angesichts dieser Raten sollte Hydroxylamin in Kulturen, die auf Nitrat wachsen, unmittelbar nach der Produktion verbraucht werden. Da Hydroxylamin mutagen ist, nehmen wir an, dass dies den früheren Selektionsdruck widerspiegelt, der darauf abzielt, während des Wachstums niedrige Hydroxylaminwerte aufrechtzuerhalten.
Es wurde zwar versucht, die Biomasse mit 15N-NH2OH in N. profundicola-Kulturen zu markieren, doch war dies bisher nicht erfolgreich (Daten nicht gezeigt). In Anbetracht der Tatsache, dass Hydroxylamin in Batch-Kulturen in niedrigen Konzentrationen im Verhältnis zum unmarkierten Nitrat zugeführt werden muss, um Toxizität zu vermeiden, und dass der überwiegende Teil des N aus Hydroxylamin in Ammonium umgewandelt werden sollte (>90 % basierend auf Abbildung 3), ist dieses Ergebnis nicht überraschend. Kurzfristige Markierungsexperimente mit viel höheren Konzentrationen von markiertem Hydroxylamin als einziger N-Quelle könnten diesen Punkt endgültig klären.
Zusammenfassend lässt sich sagen, dass die hier vorgestellten Daten mit einem Modell übereinstimmen, bei dem N. profundicola Nitrat über ein freies Hydroxylamin-Zwischenprodukt reduziert. Diese Experimente schließen die Möglichkeit noch nicht völlig aus, dass die in N. profundicola vorgeschlagene Reverse-HURM-Aktivität als eine Ammonium-bildende Nitritreduktase funktioniert, die 6 Elektronen überträgt, ohne ein N-Oxid-Zwischenprodukt freizusetzen. Unveröffentlichte Ergebnisse deuten darauf hin, dass einige Punktmutationen im nrfA-Gen die Pentaheme-Cytochrom-c-Nitrit-Reduktase zu einem undichten Enzym machen können, das N-Oxid-Zwischenprodukte freisetzt und Nitrit nicht stöchiometrisch in Ammonium umwandelt (Jörg Simon, pers. Mitt.). In Anbetracht der evolutionären Verwandtschaft zwischen NrfA und HaoA (Bergmann et al., 2005; Simon und Klotz, 2013) könnte das Reverse-HURM-Modul in N. profundicola natürlicherweise als Ammonium- und Hydroxylamin-produzierender Enzymkomplex funktionieren, während NrfA als Ammonium-produzierende Nitritreduktase „leckfrei“ arbeitet. Weitere Experimente unter Verwendung von Har/Hcp-Inhibitoren mit Nitrat oder Nitrit als Substrat in ganzen Zellen und/oder biochemische Untersuchungen des Reverse-HURM-Komplexes zur Bestimmung des In-vitro-Produkts sollten zusätzliche direkte Tests dieser Hypothese liefern. Unabhängig vom spezifischen Mechanismus deutet die umfangreiche Produktion von Ammonium, die weit über den biosynthetischen Bedarf von N. profundicola hinausgeht, darauf hin, dass es und verwandte Epsilonproteobakterien eine ökologische Rolle als Ammoniumquelle in nitratreichen, ammoniumarmen, hypoxischen Umgebungen spielen können, in denen sie häufig vorkommen.
Materialien und Methoden
Berechnungen der freien Energie
Die freien Energien von Gibbs (Tabelle 2) wurden aus den Standard-Redoxpotentialen der Halbreaktionen mit Hilfe der Nernst-Gleichung (Δ G0 = -nFE0) oder durch die Summierung der freien Energien der Halbreaktionen aus der Literatur (Thauer et al., 1977; van der Star et al., 2008). Alle Berechnungen wurden unter Standardbedingungen durchgeführt (1 M alle Arten, 25°C, pH = 7).
Kultivierung und Analyse der Stickstoffverbindungen
N. profundicola Stamm AmH (ATCC BAA-1463) wurde wie zuvor beschrieben kultiviert und die Zellen gezählt (Smith et al., 2008; Campbell et al., 2009). Das Ammonium in den Kulturüberständen wurde nach Derivatisierung mit Dansylchlorid und Trennung durch HPLC mit Fluoreszenzdetektion quantifiziert (Shakila et al., 2001). Nitrat wurde durch HPLC mit UV/Vis-Detektion quantifiziert. Hydroxylaminkonzentrationen wurden in Kulturfiltraten (0,2 μm) wie von Frear und Burrell (1955) beschrieben bestimmt.
15N-Markierungsverfahren
Stammlösungen von 20 Atom-% 15N-Nitrat und 15N-Ammonium wurden durch Mischen von >99 Atom-% und Salzen in natürlicher Häufigkeit hergestellt, um ein Medium mit einer Endkonzentration von 5 mM Nitrat oder Ammonium je nach Bedarf herzustellen. Die Zellen wurden aus den 15N-markierten Kulturen durch Zentrifugation geerntet und das Zellpellet dreimal mit nanoreinem Wasser gewaschen, um Salze zu entfernen. Die Zellsuspension wurde in Zinnkapseln getrocknet, die an das Labor für stabile Isotope der University of California-Davis geschickt wurden, wo der Anteil an 15N-Atomen durch Isotopenverhältnis-Massenspektrometrie bestimmt wurde. Kontrollproben zeigten, dass während der Probenverarbeitung keine isotopische Kontamination aufgetreten war (Daten nicht gezeigt).
Quantitative PCR
Gen-spezifische Primer (Tabelle 3) wurden zur Quantifizierung der mRNA-Häufigkeit in der aus N. profundicola extrahierten Gesamt-RNA verwendet, wie zuvor beschrieben (Campbell et al., 2009).

Tabelle 3. Primer und Bedingungen für die Q-RT-PCR-Quantifizierung spezifischer Gene in Gesamt-RNA-Proben, die aus N. profundicola extrahiert wurden.
Hydroxylamin-Aufnahme
N. profundicola wurde mit H2 und Formiat als Elektronendonatoren und Nitrat als einzigem Elektronenakzeptor bis zur späten exponentiellen Phase kultiviert. Die Zellen wurden durch Zentrifugation geerntet. Alle Transfers und Zellwaschungen wurden in einer anaeroben Kammer von Coy Laboratories durchgeführt. Die Zellen wurden mit nitratfreiem Wachstumsmedium bis zu einer Endkonzentration von ~109 Zellen ml-1 resuspendiert und 2 Stunden lang bei 45 °C unter einer Atmosphäre von 80 % H2 + 20 % CO2 bebrütet, bevor Hydroxylamin (5 mM Endkonzentration) aus einer anaerob hergestellten Stammlösung zugegeben wurde. Aliquots der Zellsuspension wurden filtriert und wie oben beschrieben auf Hydroxylamin und nach Derivatisierung mit Diethylethoxymethylenemalonat mittels HPLC auf Ammonium untersucht (Gómez-Alonso et al., 2007).
Beiträge der Autoren
Thomas E. Hanson, Barbara J. Campbell und Martin G. Klotz planten gemeinsam die Experimente und interpretierten die Ergebnisse, Thomas E. Hanson führte alle Analysen der Stickstoffverbindungen durch und bereitete die Proben für die Massenspektrometrie vor, Barbara J. Campbell züchtete alle Kulturen und führte die quantitative PCR durch, Katie M. Kalis führte die Experimente zur Aufnahme von Hydroxylamin durch, Mark A. Campbell identifizierte NAMH_1302 als ein relevantes Gen. Thomas E. Hanson verfasste die Arbeit, die anschließend von Thomas E. Hanson, Barbara J. Campbell und Martin G. Klotz überarbeitet wurde.
Erklärung zu Interessenkonflikten
Die Autoren erklären, dass die Forschung in Abwesenheit von kommerziellen oder finanziellen Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Dankesworte
Die Autoren danken Lisa Y. Stein (Univ. Alberta-Edmonton), Jenn Macalady (Penn State Univ.), David Kirchman und Matt Cottrell (Univ. Delaware) für die kritische Durchsicht der Manuskriptentwürfe, Jennifer Biddle für den Zugang zu einer anaeroben Kammer und Kathy Coyne für den Zugang zu einer HPLC. Dieses Projekt wurde unterstützt durch Zuschüsse der National Science Foundation an Barbara J. Campbell und S. Craig Cary (EF-0333203), an Barbara J. Campbell (DEB-0640414), an Barbara J. Campbell und Thomas E. Hanson (MCB-0950691) und an Martin G. Klotz (MCB-0948202/1202648), durch einen EPSCoR RII-2-Zuschuss an die University of Delaware (EPS-0814251) und durch Fördermittel der University of North Carolina für Martin G. Klotz.
Kern, M., und Simon, J. (2009). Elektronentransportketten und Bioenergetik des respiratorischen Stickstoffmetabolismus in Wolinella succinogenes und anderen Epsilonproteobakterien. Biochim. Biophys. Acta 1787, 646-656. doi: 10.1016/j.bbabio.2008.12.010
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Lindsey, G. A., and Rhines, C. M. (1932). Die Produktion von Hydroxylamin durch Reduktion von Nitraten und Nitriten durch verschiedene reine Bakterienkulturen. J. Bacteriol. 24, 489-492.
Pubmed Abstract | Pubmed Full Text
Loferer-Krossbacher, M., Klima, J., and Psenner, R. (1998). Bestimmung der bakteriellen Zelltrockenmasse durch Transmissionselektronenmikroskopie und densitometrische Bildanalyse. Appl. Environ. Microbiol. 64, 688-694.
Pubmed Abstract | Pubmed Full Text