ARGOMENTI DI BIOCHIMICA
Angoli diedri (o di torsione)
Definizioni di angolo di legame e angolo dihdrale (torsione). Angoli diedri della catena principale dei polipeptidi, φ, ψ e ω. Trame di Ramachandran.
Le conformazioni che le molecole biologiche adottano determinano le proprietà fisiche e chimiche che mostrano nei sistemi biologici. Pertanto, abbiamo bisogno di un modo per descrivere le conformazioni osservate per i tipi di molecole studiate in biochimica. L’attenzione principale in questa sezione sarà sulle conformazioni polipeptidiche, come preparazione alla comprensione delle relazioni struttura-funzione nelle proteine
Un angolo diedro – chiamato anche angolo di torsione – è definito da quattro atomi legati in sequenza. Questo è rappresentato nella figura sottostante dalla struttura A-B-C-D (secondo pannello; si noti la distinzione tra angolo di legame e angolo di torsione). Immaginate di guardare lungo il legame tra gli atomi B e C (come indicato dalla freccia). Questo è il legame centrale dei tre legami definiti. All’inizio, vediamo il legame B-C nel piano della figura, con il legame B-A che punta a sinistra e in alto fuori dal piano, e il legame C-D che punta in basso e a destra. Poi prendiamo questa struttura e cominciamo a girarla in modo da poter guardare direttamente il legame B-C (terzo pannello). Alla fine, la nostra vista è equivalente alla proiezione di Newman nell’ultimo pannello. Da questa vista, l’angolo diedro τ è l’angolo apparente sotteso da D rispetto ad A, come mostrato. Definiamo la gamma di valori degli angoli diedri come , e il valore di τ è circa -150° (La parte negativa della gamma corrisponde a quando D è in senso antiorario rispetto ad A.)
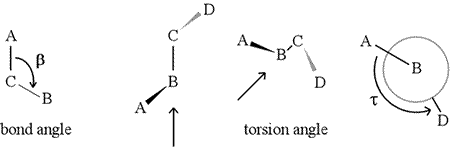
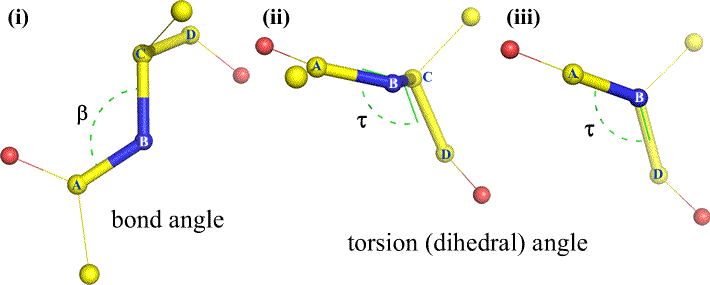
Naturalmente, altri atomi possono essere collegati a B e C – e di solito lo sono. Dovremmo specificare quali atomi corrispondono ad A, B, C e D per definire senza ambiguità l’angolo diedro. Per lunghi polimeri non ramificati, come le proteine, ha senso richiedere che da A a D siano tutti gli atomi della catena principale. Sotto c’è un’immagine di grafica molecolare che corrisponde allo schizzo di cui sopra e illustra la definizione dell’angolo diedro della catena principale del polipeptide Phi (φ).
Angoli diedri della catena principale del polipeptide: Phi (φ), Psi (ψ), e Omega (ω)
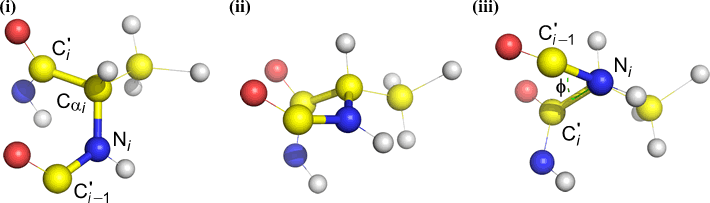
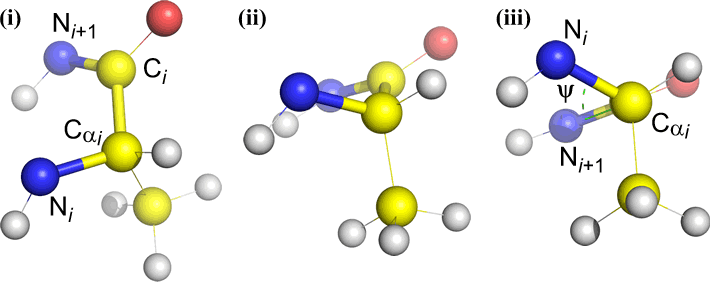
Un angolo diedro è definito da quattro atomi. Può essere visualizzato guardando il legame centrale (cioè il legame dall’atomo 2 all’atomo 3). Gli angoli diedri lungo una catena polipeptidica sono di tre tipi:
Phi (φ) – legame centrale tra N(i) e C(alfa, i). Guardando il legame N(i)-Cα(i), notare l’angolo sotteso dal carbonio acilico C(i) rispetto a C(i-1) – il carbonio acilico del residuo precedente.
Psi (ψ) – legame centrale tra C(alfa, i) e C(i) . Guarda il legame Cα(i)-C(i), nota l’angolo sotteso da N(i + 1) rispetto a N(i).
Omega (ω) – il legame peptidico tra il carbonio acile C(i) e N(i+1) è il legame centrale.
Phi (φ) e psi (ψ) sono talvolta indicati come angoli di Ramachandran, poiché sono usati in un grafico bidimensionale degli angoli diedri della catena principale chiamato grafico di Ramachandran (vedi sotto).
Ramachandran plot
Il Ramachandran plot è un grafico degli angoli diedri della catena principale phi (φ) e psi (ψ) per una catena polipeptidica, con valori phi (φ) lungo l’asse delle x e valori psi (ψ) lungo l’asse delle y.
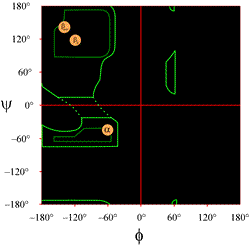
Il layout di un diagramma di Ramachandran come tipicamente presentato nei libri di testo è mostrato a sinistra (clicca sulla figura per visualizzare una versione più grande). I contorni indicano l’estensione delle combinazioni consentite (verde chiaro) e più favorite (verde scuro) di (φ, ψ). I cerchi arancioni mostrano le posizioni dei valori ideali di phi e psi per le più comuni caratteristiche strutturali secondarie regolari, i filamenti beta (in fogli antiparalleli e paralleli) e le eliche alfa. Al di fuori dei contorni, le conformazioni corrispondenti sono sfavorite o non ammesse. Le posizioni esatte dei contorni non dovrebbero essere prese troppo alla lettera, poiché le regioni favorevoli e permesse dipendono dall’identità del residuo. La glicina, in particolare, ha regioni permesse molto più grandi di quelle mostrate qui. Questo è discusso più avanti.
Un “vero” diagramma di Ramachandran
Nel “gioco finale” della determinazione della struttura delle proteine, il modello determinato sperimentalmente deve essere analizzato per la sua qualità stereochimica. Seri problemi con il modello possono essere indicati se ci sono istanze di residui non-Gly i cui angoli diedri della catena principale si trovano al di fuori delle regioni permesse, o se una proporzione troppo alta di residui è al di fuori della regione più favorita. Un esempio di un diagramma di Ramachandran per una proteina di 190 residui è mostrato nella figura allegata.
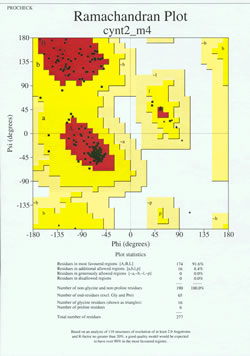
La figura mostra un diagramma di Ramachandran per una struttura proteica determinata dalla cristallografia a raggi X. (Ogni quadrato nero rappresenta la conformazione della catena principale in un residuo della proteina. Notate come i valori phi, psi per i residui si raggruppano nelle regioni rosse “più favorite”. La regione rossa in alto a sinistra (etichettata “B” nella figura) corrisponde ai residui in una conformazione beta della catena principale, mentre la grande area vicino al centro (etichettata “A”) corrisponde all’elica alfa. La terza, piccola area rossa del grafico è dove si trovano i residui che adottano un’elica alfa sinistra (“L”). Alcuni dei residui si trovano in “regioni aggiuntive permesse” (giallo brillante) che confinano con le regioni più favorite. Notate come i residui di glicina (indicati come triangoli) si trovano in alcuni casi in “regioni non permesse”. Questo perché la glicina, con solo il piccolo atomo di idrogeno come “catena laterale”, è molto meno ingombrata stericamente di altri aminoacidi. Gly è in grado di adottare coppie di angoli phi, psi che non sono permessi per nessun altro residuo.