Att härleda kopplingsavstånd och genordning från trepunktskorsningar
Då vi lägger till en tredje gen har vi nu flera olika typer av korsningsöverprodukter som kan erhållas. Följande figur visar de olika rekombinanta produkter som är möjliga.
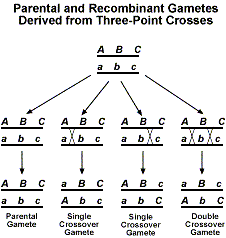
Om vi nu skulle utföra en testkorsning med F1 skulle vi förvänta oss ett 1:1:1:1:1:1:1:1:1:1:1:1:1:1:1:1-förhållande. Liksom vid tvåpunktsanalyserna som beskrivs ovan visar avvikelse från detta förväntade förhållande att koppling sker.Det bästa sättet att bekanta sig med analysen av trepunktstestkorsningsdata är att gå igenom ett exempel. Vi kommer att använda det godtyckliga exemplet med generna A, B och C. Vi gör först en korsning mellan individer som är AABBCC och aabbcc. Därefter testkryssas F1 med en individ som är aabbcc. Vi kommer att använda följande uppgifter för att bestämma genordningen och kopplingsavstånden.Liksom för tvåpunktsdata kommer vi att ta hänsyn till F1-släktkompositionen.
| Genotyp | Observerad | Typ av gamet | |
|---|---|---|---|
| ABC | Förälder | ||
| abc | Förälder | ||
| AbC | Ensam-korsning mellan generna C och B | ||
| aBc | Enkel korsning mellan generna C och B | ||
| ABc | dubbelcrossover | ||
| abC | Dubbel crossover | ||
| Abc | Enkel crossover mellan generna A och C | ||
| aBC | Enkel-crossover mellan generna A och C | ||
| Totalt |
Det bästa sättet att lösa dessa problem är att utveckla en systematisk metod.Först måste man bestämma vilka av genotyperna som är föräldragenotyper.De genotyper som förekommer mest frekvent är föräldragenotyper.Av tabellen framgår det tydligt att genotyperna ABC och abc är föräldragenotyper.
Nästan måste vi bestämma genernas ordning. När vi har bestämt föräldragenotyperna använder vi den informationen tillsammans med den information som erhållits från dubbelkorsningen. De dubbelkorsade könscellerna har alltid den lägsta frekvensen. Enligt tabellen har genotyperna ABc och abC den lägsta frekvensen. Nästa viktiga punkt är att en dubbel korsning flyttar den mellersta allelen från en systerkromatid till den andra. På så sätt placeras den icke-föräldrade allelen av den mellersta genen på en kromosom med de föräldrade allelerna av de två flankerande generna. Vi kan se i tabellen att C-genen måste vara i mitten eftersom den recessiva c-allelen nu finns på samma kromosom som A- och B-allelen, och den dominanta C-allelen finns på samma kromosom som de recessiva a- och b-allelerna.
När vi nu vet att genordningen är ACB, kan vi börja bestämma kopplingsavstånden mellan A och C och C, och C och B. Kopplingsavståndet beräknas genom att dividera det totala antalet rekombinanta könsceller med det totala antalet könsceller. Detta är samma tillvägagångssätt som vi använde för de tvåpunktsanalyser som vi utförde tidigare. Det som är annorlunda är att vi nu också måste ta hänsyn till händelser med dubbla korsningar. För dessa beräkningar inkluderar vi dessa dubbla korsningar i beräkningarna av båda intervallavstånden.
Så avståndet mellan generna A och C är 17,9 cM, och avståndet mellan C och Bis 7,0 cM .
Nu ska vi pröva ett problem från Drosophila, genom att tillämpa de principer som vi använde i exemplet ovan. Följande tabell visar de resultat som vi kommer att analysera.
| Genotyp | Observerad | Typ av gamet | |
|---|---|---|---|
| v cv+ ct+ | Förälder | ||
| v+ cv ct | Förälder | ||
| v cv ct+ | Ensam-korsning mellan generna ct och cv | ||
| v+ cv+ ct | Ensam-crossover mellan generna ct och cv | ||
| v cv ct | Enkel crossover mellan generna v och ct | ||
| v+ cv+ ct+ | Single-Crossover mellan generna v och ct | ||
| v cv+ ct | Dubbel-crossover | ||
| v+ cv ct+ | Dubbel crossover | ||
| Totalt |
Steg 1: Bestäm föräldrarnas genotyper.
De vanligaste genotyperna är de parentiska typerna. Dessa genotyper är v cv+ ct+ och v+ cv ct.Det som skiljer sig från vår första trepunktskorsning är att den ena föräldern inte innehåller alla dominanta alleler och den andra alla recessiva alleler.
Steg 2: Bestäm genordningen
För att bestämma genordningen behöver vi föräldrarnas genotyper samt genotyperna för dubbel korsning Som vi nämnde ovan är de minst frekventa genotyperna genotyperna för dubbel korsning. Dessa genotyper är v cv cv+ ct och v+ cv ct+.Utifrån denna information kan vi bestämma ordningen genom att ställa frågan:I genotyperna för dubbel korsning, vilken föräldraallel är inte associerad med de två föräldraalleler som den var associerad med i den ursprungliga föräldrakorsningen. Från den första dubbelkorsningen, v cv+ ct, är ct-allelen associerad med v- och cv+-allelerna, två alleler som den inte var associerad med i den ursprungliga korsningen. Därför är ct i mitten och genordningen är v ct cv.
Steg 3: Bestämning av kopplingsavstånden.
-
v – ct avståndsberäkning. Detta avstånd fås på följande sätt: 100*((89+94+3+5)/1448) = 13,2 cM
-
beräkning av ct – cv-avståndet. Detta avstånd beräknas på följande sätt: 100*((45+40+3+5)/1448) = 6,4 cM
Steg 4. Rita kartan.

Trepunktskorsningar gör det också möjligt att mäta interferens (I) mellan korsningshändelser inom en given region av en kromosom.Specifikt ger mängden dubbla korsningar en indikation på om interferens förekommer. Konceptet är att givet specifika rekombinationshastigheter i två intilliggande kromosomala intervaller, bör graden av dubbla korsningar i denna region vara lika med produkten av de enkla korsningarna. I exemplet v ct cv som beskrivs ovan var rekombinationsfrekvensen 0,132 mellan generna v och ct, och rekombinationsfrekvensen mellan ct och cv var 0,064. Vi skulle därför förvänta oss 0,84 % dubbla rekombinanter. Med ett urval på 1 448 personer skulle detta innebära 12 dubbla rekombinanter. I själva verket upptäckte vi endast 8.
För att mäta interferens beräknar vi först koefficienten för samverkan (c.o.c.) som är förhållandet mellan observerade och förväntade dubbla rekombinanter. Interferens beräknas sedan som 1 – c.o.c.Formeln är följande:
![]()
För v ct cvdata är interferensvärdet 33%.
För det mesta ligger interferensvärdena mellan 0 och 1. Värden som är mindre än 1 indikerar att interferens förekommer i denna region av kromosomen.