Afledning af koblingsafstand og genorden fra trepunktskrydsninger
Ved at tilføje et tredje gen har vi nu flere forskellige typer af krydsningsoverprodukter, der kan opnås. Følgende figur viser de forskellige krydsningsprodukter, der er mulige.
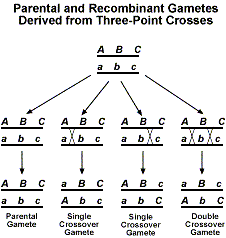
Nu, hvis vi skulle udføre et testkryds med F1, ville vi forvente et 1:1:1:1:1:1:1:1:1:1:1:1:1:1:1-forhold. Som med de topunktsanalyser, der er beskrevet ovenfor, indikerer afvigelse fra dette forventede forhold, at der forekommer sammenkobling.Den bedste måde at blive fortrolig med analysen af trepunkts testkrydsningsdata er at gennemgå et eksempel. Vi vil bruge det vilkårlige eksempel med generne A, B og C. Vi foretager først en krydsning mellem individer, der er AABBCC og aabbcc. Derefter testkrydses F1 med et individ, der er aabbcc. Vi vil bruge følgende data til at bestemme genorden og linkageafstande.Som med topunktsdataene vil vi overveje F1-gametekompositionen.
| Genotype | Observeret | Gammettype | |
|---|---|---|---|
| ABC | Forældre | ||
| abc | Forældre | ||
| AbC | Enkelt-crossover mellem generne C og B | ||
| aBc | Single-crossover mellem generne C og B | ||
| ABc | Dobbelt-crossover | ||
| abC | Dobbelt-crossover | ||
| Abc | Enkelt-crossover mellem generne A og C | ||
| aBC | Enkelt-crossover mellem generne A og C | ||
| I alt |
Den bedste måde at løse disse problemer på er ved at udvikle en systematisk fremgangsmåde.Først skal det bestemmes, hvilke af genotyperne der er forældregenotyperne.De genotyper, der findes hyppigst, er forældregenotyperne.Af tabellen fremgår det klart, at ABC- og abc-genotyperne var forældregenotyperne.
Dernæst skal vi bestemme genernes rækkefølge. Når vi har bestemt forældrenes genotyper, bruger vi disse oplysninger sammen med de oplysninger, vi har fået fra dobbeltkrydsningen. Dobbeltkrydsningsgameterne er altid i den laveste frekvens. Af tabellen fremgår det, at genotyperne ABc og abC har den laveste frekvens. Det næste vigtige punkt er, at en double-crossover-hændelse flytter den midterste allel fra den ene søsterkromatid til den anden. Det betyder, at det ikke-forældreallel af det midterste gen i praksis placeres på et kromosom sammen med forældreallelerne af de to flankerende gener. Vi kan se af tabellen, at C-genet må være i midten, fordi den recessive c-allel nu er på samme kromosom som A- og B-allelerne, og den dominerendeC-allel er på samme kromosom som de recessive a- og b-alleler.
Nu da vi ved, at genordenen er ACB, kan vi gå i gang med at bestemme linkageafstandene mellem A og C samt C og B. Linkageafstanden beregnes ved at dividere det samlede antal rekombinerede gameter med det samlede antal gameter. Dette er den samme fremgangsmåde, som vi har anvendt i forbindelse med de topunktsanalyser, som vi tidligere har foretaget. Det, der er anderledes, er, at vi nu også skal tage hensyn til de dobbelte krydsningsbegivenheder. Til disse beregninger medtager vi disse dobbelte krydsninger i beregningerne af begge intervalafstande.
Så afstanden mellem generne A og C er 17,9 cM, og afstanden mellem C og Bis 7,0 cM .
Nu skal vi prøve et problem fra Drosophila ved at anvende de principper, vi brugte i ovenstående eksempel. Følgende tabel viser de resultater, som vi vil analysere.
| Genotype | Observeret | Type af gamet | |
|---|---|---|---|
| v cv+ ct+ | Forældre | ||
| v+ cv ct | Forældre | ||
| v cv ct+ | Enkelt-krydsning mellem generne ct og cv | ||
| v+ cv+ ct | Enkelt-crossover mellem generne ct og cv | ||
| v cv ct | Enkelt-crossover mellem generne v og ct | ||
| v+ cv+ ct+ | Enkelt-crossover mellem generne v og ct | ||
| v cv+ ct | Dobbelt-crossover | ||
| v+ cv ct+ | Double-crossover | ||
| I alt |
Stræde 1: Forældregenotyperne bestemmes.
De hyppigst forekommende genotyper er de parentale typer. Disse genotyper er v cv+ ct+ og v+ cv ct.Det, der er forskelligt fra vores første trepunktskrydsning, er, at den ene forælder ikke indeholder alle de dominerende alleler og den anden alle de recessive alleler.
Stræk 2: Bestem genorden
For at bestemme genordenen har vi brug for forældrenes genotyper samt de dobbelte crossover-genotyper Som vi nævnte ovenfor, er de mindst hyppige genotyper de dobbelte crossover-genotyper. Disse genotyper er v cv cv+ ct og v+ cv ct+.Ud fra disse oplysninger kan vi bestemme rækkefølgen ved at stille spørgsmålet: Hvilken forældreallel i dobbeltkrydsningsgenotyperne er ikke associeret med de to forældrealleler, som den var associeret med i den oprindelige forældrekrydsning. Fra den første dobbeltkrydsning, v cv+ ct, er ct-allelen associeret med v- og cv+-allelerne, to alleler, som den ikke var associeret med i den oprindelige krydsning. Derfor er ct i midten, og genordenen er v ct cv.
Stræk 3: Bestemmelse af linkageafstande.
-
v – ct afstandsberegning. Denne afstand beregnes på følgende måde: 100*((89+94+3+5)/1448) = 13,2 cM
-
beregning af ct – cv-afstanden. Denne afstand beregnes på følgende måde: 100*((45+40+3+5)/1448) = 6,4 cM
Strin 4. Tegn kortet.

Trepunktskrydsninger giver også mulighed for at måle interferens(I) mellem krydsningsbegivenheder inden for et givet område af et kromosom.Specifikt giver mængden af dobbelt krydsning en indikation af, om der forekommer interferens. Konceptet er, at givet specifikke rekombinationshastigheder i to tilstødende kromosomale intervaller, skal hastigheden af dobbelte krydsninger i dette område være lig med produktet af de enkelte krydsninger. I det ovenfor beskrevne eksempel v ct cv var rekombinationsfrekvensen 0,132 mellem generne v og ct, og rekombinationsfrekvensen mellem ct og cv var 0,064. Vi ville derfor forvente 0,84% dobbelte rekombinanter. Med en stikprøvestørrelse på 1448 ville dette svare til 12 dobbelte rekombinanter. Vi påviste faktisk kun 8.
For at måle interferens beregner vi først coincidenskoefficienten (c.o.c.), som er forholdet mellem de observerede og de forventede dobbelte rekombinanter. Interferens beregnes derefter som 1 – c.o.c. Formlen er som følger:
![]()
For v ct cvdata er interferensværdien 33%.
Sædvanligvis ligger interferensværdierne mellem 0 og 1. Værdier under 1 angiver, at der forekommer interferens i dette område af kromosomet.