Derivar la distancia de enlace y el orden de los genes a partir de cruces de tres puntos
Al añadir un tercer gen, ahora tenemos varios tipos diferentes de sobreproductos de cruce que se pueden obtener. La siguiente figura muestra los diferentes productos recombinantes posibles.
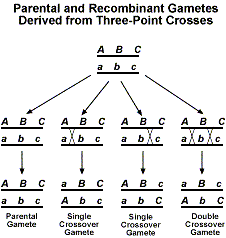
Ahora, si realizáramos un cruce de prueba con la F1, esperaríamos una proporción de 1:1:1:1:1:1:1. Al igual que con los análisis de dos puntos descritos anteriormente, la desviación de esta proporción esperada indica que se está produciendo un ligamiento.La mejor manera de familiarizarse con el análisis de los datos de los cruces de prueba de tres puntos es a través de un ejemplo. Utilizaremos el ejemplo arbitrario de los genes A, B y C. Primero hacemos un cruce entre individuos que son AABBCC y aabbcc. A continuación, el F1 se cruza con un individuo que es aabbcc. Utilizaremos los siguientes datos para determinar el orden de los genes y las distancias de enlace.Al igual que con los datos de dos puntos, consideraremos la composición de los gametos F1.
| Genotipo | Observado | Tipo de gameto |
|---|---|---|
| ABC | Parental | |
| abc | Parental | |
| AbC | Simple-cruce entre los genes C y B | |
| aBc | Cruce simple entre los genes C y B | |
| ABc | Doble-cruce | |
| abC | Cruce doble | |
| Abc | Cruce simple entre los genes A y C | |
| aBC | .cruce entre los genes A y C | |
| Total |
La mejor manera de resolver estos problemas es desarrollar un enfoque sistemático.En primer lugar, hay que determinar cuáles son los genotipos parentales.Los genotipos encontrados con más frecuencia son los genotipos parentales.De la tabla se desprende que los genotipos ABC y abc son los genotipos parentales.
A continuación hay que determinar el orden de los genes. Una vez que hayamos determinado los genotipos parentales, utilizaremos esa información junto con la obtenida en el cruce doble. Los gametos de doble cruce son siempre los de menor frecuencia. En la tabla, los genotipos ABc y abC son los de menor frecuencia. El siguiente punto importante es que un evento de doble cruce mueve el alelo medio de una cromátida hermana a la otra. Esto coloca el alelo no parental del gen del medio en un cromosoma con los alelos parentales de los dos genes que lo flanquean. Podemos ver en la tabla que el gen C debe estar en el medio porque el alelo recesivo C está ahora en el mismo cromosoma que los alelos A y B, y el alelo dominante C está en el mismo cromosoma que los alelos recesivos A y B.
Ahora que sabemos que el orden de los genes es ACB, podemos determinar las distancias de enlace entre A y C, y C y B. La distancia de enlace se calcula dividiendo el número total de gametos recombinantes entre el número total de gametos. Este es el mismo enfoque que utilizamos con los análisis de dos puntos que realizamos anteriormente. Lo que es diferente es que ahora debemos considerar también los eventos de doble cruce. Para estos cálculos incluimos esos cruces dobles en los cálculos de ambas distancias de intervalo.
Así que la distancia entre los genes A y C es de 17,9 cM, y la distancia entre C y Bis de 7,0 cM.
Intentemos ahora un problema de Drosophila, aplicando los principios que utilizamos en el ejemplo anterior. La siguiente tabla muestra los resultados que analizaremos.
| Genotipo | Observado | Tipo de gameto |
|---|---|---|
| v cv+ ct+ | Parental | |
| v+ cv ct | Parental | |
| v cv ct+ | Single-cruce entre los genes ct y cv | |
| v+ cv+ ct | Simple-cruce entre los genes ct y cv | |
| v cv ct | Cruce simple entre los genes v y ct | |
| v+ cv+ ct+ | Single-cruce entre genes v y ct | |
| v cv+ ct | Doble-cruce | |
| v+ cv ct+ | Doble cruce | |
| Total |
Paso 1: Determinar los genotipos parentales.
Los genotipos más abundantes son los tipos parentales. Estos genotipos son v cv+ ct+ y v+ cv ct.Lo que difiere de nuestro primer cruce de tres puntos es que uno de los padres no contenía todos los alelos dominantes y el otro todos los alelos recesivos.
Paso 2: Determinar el orden de los genes
Para determinar el orden de los genes, necesitamos los genotipos parentales, así como los genotipos de doble cruce Como hemos mencionado anteriormente, los genotipos menos frecuentes son los genotipos de doble cruce. Estos genotipos son v cv+ ct y v+ cv ct+. A partir de esta información podemos determinar el orden planteando la siguiente pregunta: En los genotipos del doble cruce, ¿qué alelo parental no está asociado con los dos alelos parentales con los que estaba asociado en el cruce parental original? En el primer cruce doble, v cv+ ct, el alelo ct está asociado con los alelos v y cv+, dos alelos con los que no estaba asociado en el cruce original. Por lo tanto, ct está en el centro, y el orden de los genes es v ct cv.
Paso 3: Determinación de las distancias de ligamiento.
-
Caluculación de la distancia v – ct. Esta distancia se obtiene de la siguiente manera: 100*((89+94+3+5)/1448) = 13,2 cM
-
cálculo de la distancia ct – cv. Esta distancia se obtiene de la siguiente manera: 100*((45+40+3+5)/1448) = 6,4 cM
Paso 4. Dibujar el mapa.

Los cruces de tres puntos también permiten medir la interferencia (I) entre los eventos de cruce dentro de una región determinada de un cromosoma.Específicamente, la cantidad de cruces dobles da una indicación de si hay interferencia. El concepto es que, dadas las tasas de recombinación específicas en dos intervalos cromosómicos adyacentes, la tasa de cruces dobles en esta región debería ser igual al producto de los cruces simples. En el ejemplo v ct cv descrito anteriormente, la frecuencia de recombinación fue de 0,132 entre los genes v y ct, y la frecuencia de recombinación entre ct y cv fue de 0,064. Por lo tanto, cabría esperar un 0,84% de recombinantes dobles. Con un tamaño de muestra de 1.448, esto equivaldría a 12 recombinantes dobles. En realidad, sólo detectamos 8.
Para medir la interferencia, primero calculamos el coeficiente de coincidencia (c.o.c.), que es la relación entre los recombinantes dobles observados y los esperados. A continuación, se calcula la interferencia como 1 – c.o.c. La fórmula es la siguiente:
![]()
Para los datos v ct c, el valor de la interferencia es del 33%.
La mayoría de las veces, los valores de la interferencia se sitúan entre 0 y 1. Los valores inferiores a uno indican que la interferencia se produce en esta región del cromosoma.