Derivando Distância de Ligação e Ordem de Gene de Cruzamentos de Três Pontos
Adicionando um terceiro gene, temos agora vários tipos diferentes de sobreprodutos de cruzamento que podem ser obtidos. A figura seguinte mostra os diferentes produtos de cruzamento que são possíveis.
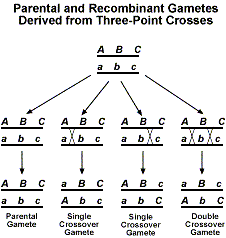
Agora, se fôssemos realizar um cruzamento de teste com F1, esperaríamos uma relação 1:1:1:1:1:1:1:1. Como nas análises de dois pontos descritas acima, o desvio desta relação esperada indica que a ligação está ocorrendo. A melhor maneira de se familiarizar com a análise dos dados de teste cruzado de três pontos é passar por um exemplo. Vamos usar o exemplo arbitrário dos genes A, B e C. Primeiro fazemos um cruzamento entre os indivíduos que são AABBCC e aabbcc. Em seguida oF1 é testado transversalmente para um indivíduo que é aabbcc. Utilizaremos os seguintes dados para determinar a ordem dos genes e as distâncias de ligação. Como com os dados de dois pontos, consideraremos a composição do jogo F1.
| Genótipo | Observado | Tipo de Gamete |
|---|---|---|
| ABC | Parental | |
| abc | > Parental | |
| AbC | > Simples-Cruzamento entre os genes C e B | |
| aBc | Cruzamento entre os genes C e B | |
| ABc | > Duplo…crossover | |
| abC | > Duplo crossover | |
| Abc | Cruzamento simples entre genes A e C | |
| aBC | Simples-cruzamento entre os genes A e C | |
| Total |
A melhor maneira de resolver estes problemas é desenvolver uma abordagem sistemática.Primeiro, determinar quais dos genótipos são os gentotipos parentais. Os genótipos encontrados com mais freqüência são os genótipos parentais. Da tabela está claro que os genótipos ABC e abc eram os genótipos parentais.
Próximo precisamos determinar a ordem dos genes. Uma vez determinados os genótipos parentais, usamos essa informação juntamente com a informação obtida a partir do cruzamento duplo. Os gametas de cruzamento duplo estão sempre na frequência mais baixa. Da tabela, os genótipos ABcand abC estão na frequência mais baixa. O próximo ponto importante é que um evento de cruzamento duplo move o alelo médio de um sisterchromatid para o outro. Isso efetivamente coloca o alelo não parental do gene médio em um cromossomo com os alelos parentais dos dois genes de flanco. Podemos ver na tabela que o Cgene deve estar no meio porque o alelo C recessivo está agora no mesmo cromossoma que os alelos A e B, e o alelo C dominante está no mesmo cromossoma que o recessivo a e os alelos balés.
Agora que sabemos que a ordem do gene é ACB, podemos determinar as distâncias de ligação entre A e C, e C e B. A distância de ligação é calculada dividindo o número total de gametas recombinantes no número total de gametas. Esta é a mesma abordagem que usamos com as análises de dois pontos que realizamos anteriormente. O que é diferente é que agora também temos de considerar os eventos de crossover duplo. Para estes cálculos nós incluímos os eventos de cruzamento duplo nos cálculos de ambas as distâncias de intervalo.
Então a distância entre os genes A e C é 17,9 cM, e a distância entre C e Bis 7,0 cM .
Agora vamos tentar um problema de Drosophila, aplicando os princípios que usamos no exemplo acima. A tabela seguinte dá os resultados que vamos analisar.
| Genótipo | Observado | Tipo de Gamete | |
|---|---|---|---|
| v cv+ ct+ | Parental | ||
| v+ cv ct | Parental | ||
| v cv ct+ | Simples-cruzamento entre os genes ct e cv | ||
| v+ cv+ ct | Simples-cruzamento entre os genes ct e cv | ||
| v cv ct | > Cruzamento simples entre genes v e ct | ||
| v+ cv+ ct+ | cruzamento entre genes v e ct | ||
| v cv+ ct | Duplo…crossover | ||
| v+ cv ct+ | Duplo crossover | ||
| Total |
Passo 1: Determinar os genótipos parentais.
Os genótipos mais abundantes são os tipos partenais. Estes genótipos são v cv+ ct+ e v+ cv ct.O que é diferente do nosso primeiro cruzamento de três pontos é que um parênteses não contém todos os alelos dominantes e o outro todos os alelos recessivos.
Passo 2: Determinar a ordem dos genes
Para determinar a ordem dos genes, precisamos dos genótipos parentais, bem como dos genótipos cruzados duplos Como mencionamos acima, os genótipos menos frequentes são os genótipos cruzados duplos. Estes genótipos sãov cv+ ct e v+ cv ct+. A partir desta informação podemos determinar a ordem fazendo a pergunta:Nos genótipos de cruzamento duplo, qual alelo parental não está associado com os dois alelos parentais aos quais foi associado no cruzamento parental original. Do primeiro cruzamento duplo, v cv+ ct, o alelo ct está associado aos alelos v andcv+, dois alelos aos quais não estava associado no cruzamento original. Portanto, ct está no meio, e a ordem gênica é v ct cv.
Passo 3: Determinação das distâncias de ligação.
-
v – caluculação da distância ct. Esta distância é derivada da seguinte forma: 100*((89+94+3+5)/1448) = 13.2 cM
-
ct – cálculo da distância cv. Esta distância é derivada da seguinte forma: 100*((45+40+3+5)/1448) = 6,4 cM
Passo 4. Desenhe o mapa.

Três pontos de cruzamento também permitem medir a interferência(I) entre eventos de cruzamento dentro de uma determinada região de um cromossoma. O conceito é que dada a taxa de recombinação específica em dois intervalos cromossômicos adjacentes, a taxa de cruzamento duplo nesta região deve ser igual ao produto do cruzamento simples. No exemplo v ct cv descrito acima, a frequência de recombinação foi de 0,132 entre os genes v e ct, e a frequência de recombinação entre ct e cv foi de 0,064. Portanto, esperaríamos 0,84% de recombinantes duplos. Com o tamanho da amostra de 1448, isto equivaleria a 12 recombinantes duplos. A interferência é então calculada como 1 – c.o.c.A fórmula é a seguinte:
![]()
Para os cvdados v ct, o valor da interferência é 33%.
A maioria das vezes, os valores da interferência caem entre 0 e 1. Valores inferiores a um indicam que a interferência está ocorrendo nesta região do cromossoma.