Ableitung der Kopplungsdistanz und der Genreihenfolge aus Dreipunktkreuzungen
Durch Hinzufügen eines dritten Gens haben wir nun mehrere verschiedene Arten von Kreuzungsüberprodukten, die erhalten werden können. Die folgende Abbildung zeigt die verschiedenen möglichen Rekombinantenprodukte.
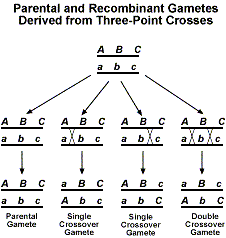
Wenn wir nun eine Testkreuzung mit F1 durchführen würden, würden wir ein Verhältnis von 1:1:1:1:1:1:1:1:1 erwarten. Wie bei den oben beschriebenen Zwei-Punkt-Analysen zeigt eine Abweichung von diesem erwarteten Verhältnis an, dass eine Kopplung vorliegt. Wir verwenden das willkürliche Beispiel der Gene A, B und C. Wir machen zunächst eine Kreuzung zwischen Individuen, die AABBCC und aabbcc sind. Dann wird dasF1 testweise mit einem Individuum gekreuzt, das aabbcc ist. Wir verwenden die folgenden Daten, um die Genreihenfolge und die Bindungsabstände zu bestimmen, wobei wir, wie bei den Zweipunktdaten, die F1-Gameten-Zusammensetzung berücksichtigen.
| Genotyp | Beobachtetes | Typ der Gamete |
|---|---|---|
| ABC | Elternteil | |
| abc | Elternteil | |
| AbC | Einzel-Crossover zwischen den Genen C und B | |
| aBc | Einfach-Crossover zwischen den Genen C und B | |
| ABc | Doppel-Crossover | |
| abC | Doppel-Crossover | |
| Abc | Einfach-Crossover zwischen Genen A und C | |
| aBC | Einfach-Crossover zwischen den Genen A und C | |
| Gesamt |
Die beste Art, diese Probleme zu lösen, besteht darin, einen systematischen Ansatz zu entwickeln.Die am häufigsten gefundenen Genotypen sind die elterlichen Genotypen.
Aus der Tabelle geht hervor, dass die Genotypen ABC und abc die elterlichen Genotypen sind.
Als nächstes müssen wir die Reihenfolge der Gene bestimmen. Nachdem wir die elterlichen Genotypen bestimmt haben, verwenden wir diese Informationen zusammen mit den Informationen, die wir durch die Doppelkreuzung erhalten haben. Die Gameten mit doppelter Kreuzung sind immer in der niedrigsten Häufigkeit vorhanden. Aus der Tabelle geht hervor, dass die Genotypen ABc und abC die geringste Häufigkeit aufweisen. Der nächste wichtige Punkt ist, dass ein Doppelkreuzungsereignis das mittlere Allel von einem Schwesterchromatid zum anderen verschiebt. Dadurch wird das nicht elterliche Allel des mittleren Gens auf ein Chromosom mit den elterlichen Allelen der beiden flankierenden Gene übertragen. Aus der Tabelle geht hervor, dass sich das C-Gen in der Mitte befinden muss, da sich das rezessive C-Allel nun auf demselben Chromosom befindet wie die A- und B-Allele und das dominante C-Allel auf demselben Chromosom wie die rezessiven A- und B-Allele.
Nun, da wir wissen, dass die Genreihenfolge ACB ist, können wir die Kopplungsabstände zwischen A und C sowie C und B bestimmen. Der Bindungsabstand wird berechnet, indem die Gesamtzahl der rekombinanten Gameten durch die Gesamtzahl der Gameten geteilt wird. Dies ist derselbe Ansatz, den wir bei den zuvor durchgeführten Zwei-Punkt-Analysen verwendet haben. Der Unterschied besteht darin, dass wir jetzt auch die Doppelkreuzungsereignisse berücksichtigen müssen. Für diese Berechnungen beziehen wir diese Doppelkreuzungen in die Berechnungen der beiden Intervallabstände ein.
Der Abstand zwischen den Genen A und C beträgt also 17,9 cM, der Abstand zwischen C und Bis 7,0 cM.
Wir wollen nun ein Problem von Drosophila versuchen, indem wir die Prinzipien anwenden, die wir im obigen Beispiel verwendet haben. Die folgende Tabelle zeigt die Ergebnisse, die wir analysieren werden.
| Genotyp | Beobachtet | Typ der Gamete |
|---|---|---|
| v cv+ ct+ | Parental | |
| v+ cv ct | Parental | |
| v cv ct+ | Single-Kreuzung zwischen den Genen ct und cv | |
| v+ cv+ ct | Einzel-Crossover zwischen den Genen ct und cv | |
| v cv ct | Single-Crossover zwischen den Genen v und ct | |
| v+ cv+ ct+ | Single-Kreuzung zwischen den Genen v und ct | |
| v cv+ ct | Doppel-Kreuzung | |
| v+ cv ct+ | Doppel-Kreuzung | |
| Insgesamt |
Schritt 1: Bestimmen Sie die elterlichen Genotypen.
Die häufigsten Genotypen sind die elterlichen Typen. Diese Genotypen sind v cv+ ct+ und v+ cv ct.Der Unterschied zu unserer ersten Drei-Punkt-Kreuzung besteht darin, dass ein Elternteil nicht alle dominanten Allele und der andere nicht alle rezessiven Allele enthält.
Schritt 2: Bestimmung der Genreihenfolge
Um die Genreihenfolge zu bestimmen, benötigen wir die elterlichen Genotypen sowie die Genotypen der doppelten Kreuzung Wie bereits erwähnt, sind die am wenigsten häufigen Genotypen die Genotypen der doppelten Kreuzung. Diese Genotypen sindv cv+ ct und v+ cv ct+.Anhand dieser Informationen können wir die Reihenfolge bestimmen, indem wir die Frage stellen: Welches elterliche Allel ist bei den Genotypen der Doppelkreuzung nicht mit den beiden elterlichen Allelen assoziiert, mit denen es bei der ursprünglichen elterlichen Kreuzung assoziiert war? Bei der ersten Doppelkreuzung, v cv+ ct, ist das Allel ct mit den Allelen v undcv+ assoziiert, zwei Allelen, mit denen es bei der ursprünglichen Kreuzung nicht assoziiert war. Daher befindet sich ct in der Mitte, und die Genfolge ist v ct cv.
Schritt 3: Bestimmung der Bindungsabstände.
-
Berechnung des Abstandes v – ct. Dieser Abstand wird wie folgt abgeleitet: 100*((89+94+3+5)/1448) = 13.2 cM
-
ct – cv Abstandsberechnung. Dieser Abstand wird wie folgt abgeleitet: 100*((45+40+3+5)/1448) = 6,4 cM
Schritt 4. Zeichnen Sie die Karte.

Die Drei-Punkt-Kreuzungen ermöglichen es auch, die Interferenz (I) zwischen den Crossover-Ereignissen innerhalb einer bestimmten Region eines Chromosoms zu messen. Das Konzept besteht darin, dass bei bestimmten Rekombinationsraten in zwei benachbarten chromosomalen Intervallen die Rate der Doppelkreuzungen in dieser Region gleich dem Produkt der Einzelkreuzungen sein sollte. In dem oben beschriebenen Beispiel v ct cv lag die Rekombinationshäufigkeit zwischen den Genen v und ct bei 0,132 und die Rekombinationshäufigkeit zwischen ct und cv bei 0,064. Daher würden wir 0,84 % Doppelrekombinanten erwarten. Bei einer Stichprobengröße von 1448 würde dies 12 Doppelrekombinanten bedeuten. Tatsächlich wurden nur 8 entdeckt.
Um die Interferenz zu messen, berechnen wir zunächst den Koinzidenzkoeffizienten (c.o.c.), der das Verhältnis zwischen beobachteten und erwarteten Doppelrekombinanten darstellt. Die Interferenz wird dann als 1 – c.o.c. berechnet. Die Formel lautet wie folgt:
![]()
Für die v ct cvdata beträgt der Interferenzwert 33%.
Meistens liegen die Interferenzwerte zwischen 0 und 1. Werte unter 1 zeigen an, dass in diesem Bereich des Chromosoms Interferenz auftritt.