Deriving Linkage Distance and Gene Order From Three-Point Crosses
Dodając trzeci gen, mamy teraz kilka różnych typów krzyżowania nadproduktów, które można uzyskać. Poniższy rysunek pokazuje różne produkty rekombinacji, które są możliwe.
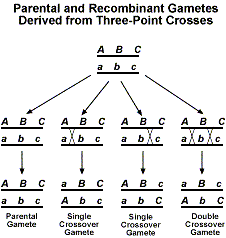
Teraz, gdybyśmy mieli wykonać krzyżówkę testową z F1, oczekiwalibyśmy stosunku 1:1:1:1:1:1:1:1:1. Podobnie jak w przypadku analizy dwupunktowej opisanej powyżej, odchylenie od tego oczekiwanego stosunku wskazuje, że występuje powiązanie.Najlepszym sposobem na zapoznanie się z analizą danych z krzyżowania trzypunktowego jest przejście przez przykład. Użyjemy arbitralnego przykładu genów A, B i C. Najpierw wykonujemy krzyżówkę pomiędzy osobnikami, które są AABBCC i aabbcc. Następnie F1 jest testcrossed do osobnika, który jest aabbcc. Do określenia kolejności genów i odległości między nimi wykorzystamy następujące dane. Podobnie jak w przypadku danych dwupunktowych, rozważymy skład gamety F1.
| Genotyp | Obserwowany | Typ gamety | |
|---|---|---|---|
| ABC | . Rodzicielskie | ||
| abc | Rodzicielskie | ||
| AbC | Pojedyncze-.crossover pomiędzy genami C i B | ||
| aBc | Single-crossover pomiędzy genami C i B | ||
| ABc | Double-crossover | ||
| abC | Double-crossover | ||
| Abc | Single-crossover między genami A i C | ||
| aBC | Single-crossover między genami A i C | ||
| Razem |
Najlepszym sposobem rozwiązania tych problemów jest opracowanie systematycznego podejścia.Po pierwsze, należy określić, które z genotypów są genotypami rodzicielskimi. Genotypy występujące najczęściej są genotypami rodzicielskimi. Z tabeli jasno wynika, że genotypy ABC i abc są genotypami rodzicielskimi.
Następnie musimy określić kolejność genów. Po określeniu genotypów rodzicielskich, używamy tej informacji wraz z informacją uzyskaną z podwójnej krzyżówki. Gamety z podwójnej krzyżówki mają zawsze najniższą częstotliwość. Z tabeli wynika, że genotypy ABc i abC mają najniższą częstotliwość. Następnym ważnym punktem jest to, że zdarzenie podwójnej krzyżówki przenosi środkowy allel z jednego chromatydu siostrzanego do drugiego. To efektywnie umieszcza nierodzicielski allel środkowego genu na chromosomie z rodzicielskimi allelami dwóch genów flankujących. Widzimy z tabeli, że gen C musi być w środku, ponieważ recesywny allel c znajduje się teraz na tym samym chromosomie co allele A i B, a dominujący allel C jest na tym samym chromosomie co recesywne allele a i balle.
Teraz, gdy wiemy, że kolejność genów to ACB, możemy zająć się określeniem odległości powiązań między A i C oraz C iB. Odległość pokrewieństwa jest obliczana przez podzielenie całkowitej liczby zrekombinowanych gamet na całkowitą liczbę gamet. Jest to takie samo podejście, jakie zastosowaliśmy w przypadku analiz dwupunktowych, które przeprowadziliśmy wcześniej. Różnica polega na tym, że musimy teraz uwzględnić również przypadki podwójnego krzyżowania. Dla tych obliczeń uwzględniamy te podwójne crossing-over w obliczeniach obu odległości interwałowych.
Więc odległość między genami A i C wynosi 17.9 cM, a odległość między C i Bis 7.0 cM .
Spróbujmy teraz problem z Drosophila, stosując zasady, których użyliśmy w powyższym przykładzie. Poniższa tabela przedstawia wyniki, które będziemy analizować.
| Genotyp | Obserwowany | Typ gamety |
|---|---|---|
| v cv+ ct+ | . Rodzicielskie | |
| v+ cv ct | Rodzicielskie | |
| v cv ct+ | Pojedyncze-.krzyżówka między genami ct i cv | |
| v+ cv+ ct | Pojedyncze-crossover pomiędzy genami ct i cv | |
| v cv ct | Single- crossover pomiędzy genami v i ct | |
| v+ cv+ ct+ | Single-krzyżówka między genami v i ct | |
| v cv+ ct | Podwójna-crossover | |
| v+ cv ct+ | Double-crossover | |
| Total |
Krok 1: Określenie genotypów rodziców.
Najbardziej obfitymi genotypami są typy rodzicielskie. Te genotypy to v cv+ ct+ i v+ cv ct.To, co różni nas od naszej pierwszej krzyżówki trzypunktowej, to fakt, że jeden z rodziców nie zawierał wszystkich alleli dominujących, a drugi wszystkich alleli recesywnych.
Krok 2: Określenie kolejności genów
Aby określić kolejność genów, potrzebujemy genotypów rodzicielskich, a także genotypów podwójnej krzyżówkiJak już wspomnieliśmy, najrzadziej występującymi genotypami są genotypy podwójnej krzyżówki. Te genotypy tov cv+ ct i v+ cv ct+. Na podstawie tych informacji możemy określić kolejność, zadając pytanie: W genotypach podwójnej krzyżówki, który allel rodzicielski nie jest związany z dwoma allelami rodzicielskimi, z którymi był związany w pierwotnej krzyżówce rodzicielskiej. Z pierwszej podwójnej krzyżówki, v cv+ ct, allel ct jest związany z allelami v i cv+, dwoma allelami, z którymi nie był związany w oryginalnej krzyżówce. Dlatego ct jest w środku, a rząd genów to v ct cv.
Krok 3: Określenie odległości pokrewieństwa.
-
kalkulacja odległości v – ct. Odległość tę uzyskuje się w następujący sposób: 100*((89+94+3+5)/1448) = 13,2 cM
-
obliczenie odległości ct – cv. Odległość tę uzyskuje się w następujący sposób: 100*((45+40+3+5)/1448) = 6,4 cM
Krok 4. Narysuj mapę.

Trzypunktowe krzyżowanie pozwala również na pomiar interferencji (I) wśród zdarzeń krzyżowania w danym regionie chromosomu.Konkretnie, ilość podwójnego krzyżowania daje wskazówkę, czy występuje interferencja. Koncepcja polega na tym, że biorąc pod uwagę określone współczynniki rekombinacji w dwóch sąsiednich przedziałach chromosomalnych, współczynnik podwójnych krzyżówek w tym regionie powinien być równy iloczynowi pojedynczych krzyżówek. W opisanym powyżej przykładzie v ct cv częstość rekombinacji między genami v i ct wynosiła 0,132, a częstość rekombinacji między ct i cv wynosiła 0,064. Dlatego spodziewamy się 0,84% podwójnych rekombinantów. Przy liczebności próby 1448, wyniosłoby to 12 podwójnych rekombinantów. My w rzeczywistości wykryliśmy tylko 8.
Aby zmierzyć interferencję, najpierw obliczamy współczynnik koincydencji (c.o.c.), który jest stosunkiem obserwowanych do oczekiwanych podwójnych rekombinantów. Interferencja jest następnie obliczana jako 1 – c.o.c.Wzór jest następujący:
![]()
Dla danych v ct cvdata, wartość interferencji wynosi 33%.
Najczęściej wartości interferencji mieszczą się w przedziale od 0 do 1. Wartości mniejsze niż jeden wskazują, że interferencja występuje w tym regionie chromosomu.