 |
 |
|
| Download | OSX 10.12+ | |
| 上のアイコンをクリックして、最新のApE (v2.0.61, Februrary 5, 2020)をダウンロードしてください。Macにオープンソースをインストールするには、以下の手順を参照してください。 OSX El Capitain (OSX 10.11) またはそれ以前のシステムでインストールする場合。 | ||
Click here  to make a voluntary donation in support of ApE. to make a voluntary donation in support of ApE. |
Follow @ApEplasmid | |
ApE install on a Mac:
Your browser does not support the <video> tag.
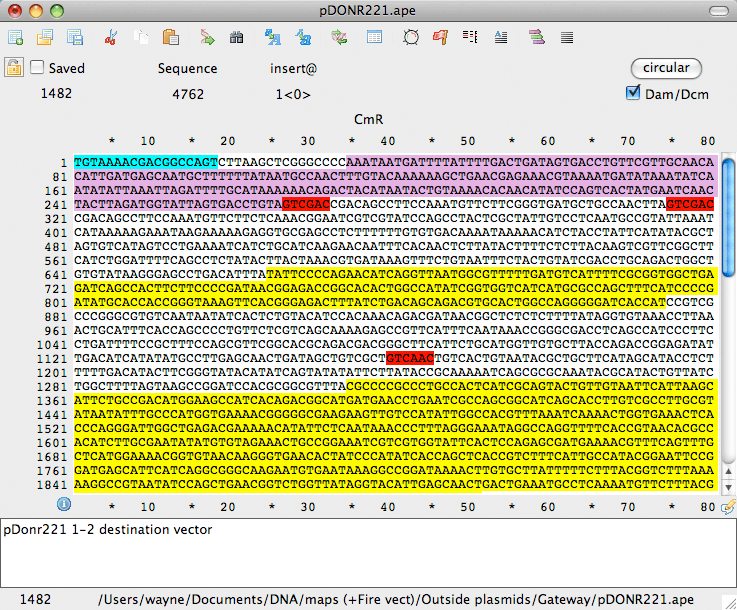 |
Windows (XP, Vista, 7, 8, 10) および Mac (OS X v10.) で実行可能です。5 以上) 制限部位を編集ウィンドウでハイライト 酵素部位の Dam/Dcm ブロッキングを正確に反映 定義済みおよびカスタム機能ライブラリを使用したテキストのハイライト 選択した DNA の翻訳、Tm、%GC、ORF をリアルタイムで読み取る DNA Strider、Fasta, GenbankおよびEMBLファイル GenbankおよびEMBLファイルから、DNA Strider互換またはGenbankファイルフォーマットとしてファイルを保存 GenbankおよびEMBLファイルからのフィーチャーアノテーションを用いて、グラフィックマップをハイライトおよび描画 NCBIまたはwormbaseで選択した配列を直接BLAST |
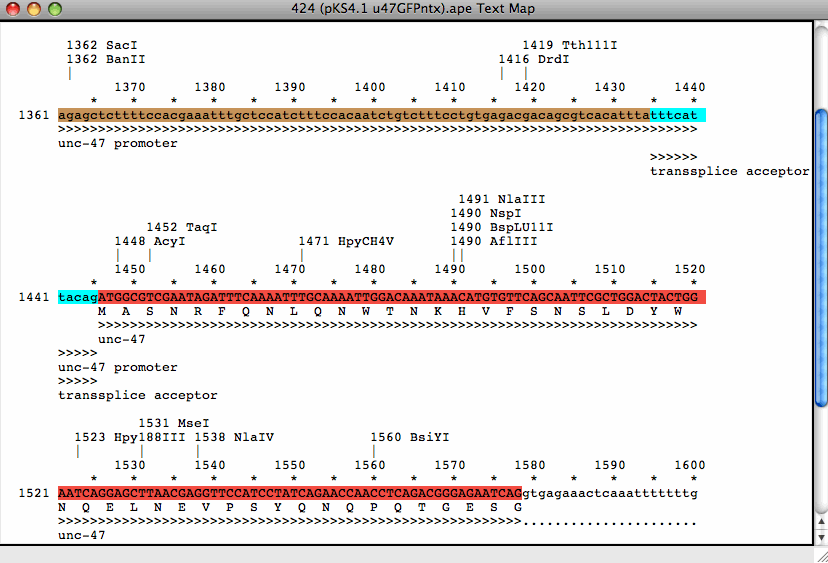
テキストマップにはDNA配列、翻訳が含まれます。 と特徴をテキストベースのグラフィックスで表示 |
 |
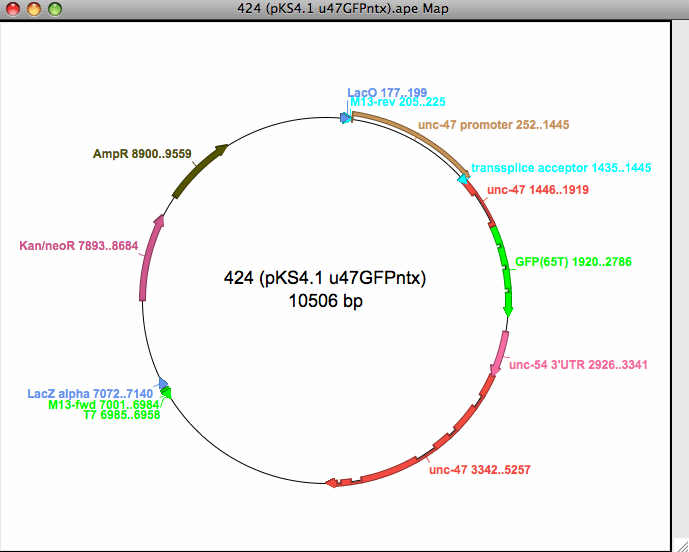 |
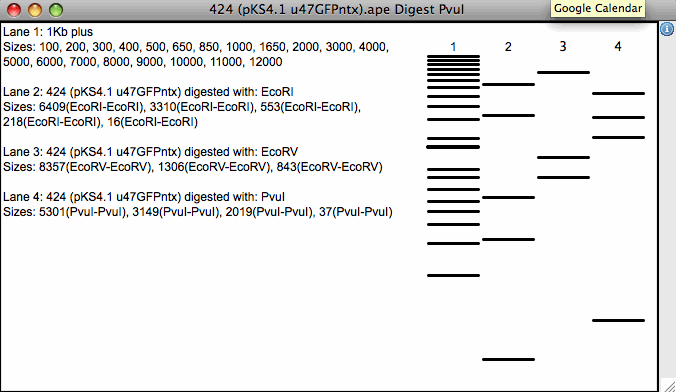
グラフィック制限マップを作成-。 グラフィックとテキストをハイパーリンクのダブルクリックで接続。 グラフィックをカプセル化されたポストスクリプトまたはスケーラブルベクターグラフィックとして保存。定義済みおよびユーザー定義の DNA ラダー バンドとテキストをダブルクリックで接続 |
 |
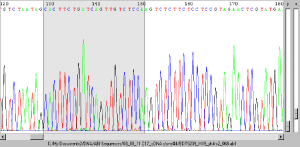 |
Reads ABI シークエンス・トレースファイル ABI トレースにある配列は直接参照配列に整合することが可能です。 アライメントがハイパーリンクされ、トレースに戻ってくる。 |
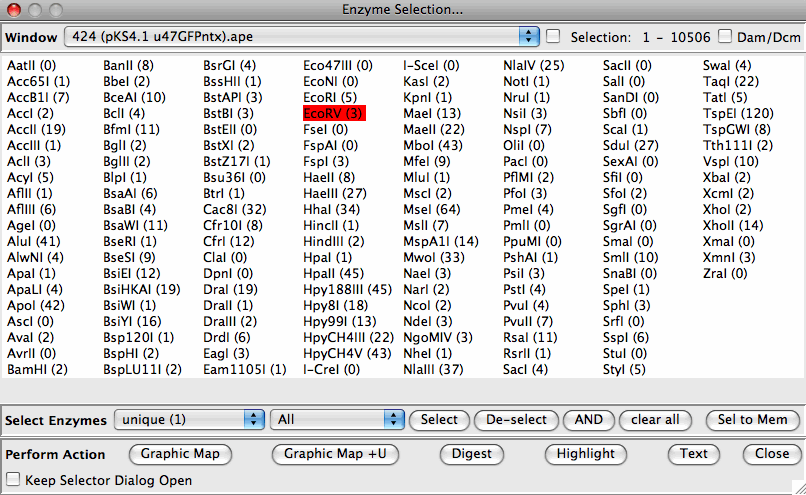
すべてのウィンドウで複数の条件(結合/交差-カット頻度、サイトタイプ)に一致するサイトを選択します。 ある配列で他の配列よりも頻繁にカットするサイト(Snip-SNP検出や診断消化のために)を選択します。 名前と認識部位で新しい酵素を定義可能 REBASEから利用可能なDNA Striderフォーマットファイル(シンプルな酵素、部位リスト)をインポート |
 |
その他の機能: |
ほとんどの解析ウィンドウには該当する配列へのリンクが張られている、以下を参照せよ。 Graphic Maps Text maps Virtual Digests Alignments (including ABI sequences) Silent Sites Translation Primer Find カスタム機能定義ライブラリの使用により、以下のことが可能になります。 配列の迅速な注釈付け 配列にハイブリダイズする、自分(または他の人)が持っているすべての利用可能なプライマーの迅速な検索と強調表示 配列の注釈付けと可視化を、複数の方法で迅速かつ効率的に行う プライマー結合部位とすべての興味深い配列特徴を示すグラフィックマップ DNAアライメントのオプションで配列を翻訳 ユーザー基準に一致するプライマーの候補を検索する(長さ。 Tm, %GC, 自己/他者相補性) 2つのDNA配列(または配列とABIトレースの任意の組み合わせ)をアラインメントし、アラインメントは元の配列にハイパーリンクされます 翻訳抑制部位を見つける グラフィック ORFマップを描く |
ApE支援のための任意の寄付をする場合はこちら をクリックします。
をクリックします。
Get older versions
Wormbaseからゲノム領域を直接エクスポートすることができるようになりました。 右上のドロップダウンメニューボタンで、”Download Track Data “を選択します。 Configure… “ボタンをクリックします。 バージョン3 Across currently visible region を使って選択したフィーチャーをダンプします。 Save to File にチェックを入れる。 Embed DNA sequence “をチェックする。 Include track configuration data “のチェックをはずします。 Go “をクリックする。 .gff ファイルを最新の ApE で開きます。
また、ゲノムビューアからゲノム領域を FASTA 形式でエクスポートすることもできます(左上のメニューから)。 同じメニューから GFF3 フィーチャートラックファイルをダウンロードすることで、フィーチャートラックを追加できます。 ApE で FASTA ファイルを開き、Features メニューで GFF3 トラック情報を開きます。
もう一つの方法は、(遺伝子ページから)遺伝子モデルを取り出し、ApE ウィンドウに貼り付けて、すべて選択し、新しいフィーチャー(Feature メニュー)を作り、現れたフィーチャー編集ウィンドウで “upper case only” ボタンを押すことです。
配列中に存在するとわかっているClaIサイト(またはXbaIやBclI)がApEですべて見つからない場合は、配列のDam/Dcmメチル化をオフにしてもう一度試してみてください。
ApEを支援するための任意の寄付は、こちら をご覧ください。
をご覧ください。
最終更新日 2020年7月3日