Derivare la distanza di linkage e l’ordine dei geni dagli incroci a tre punti
Aggiungendo un terzo gene, ora abbiamo diversi tipi di sovrapprodotti di incrocio che possono essere ottenuti. La figura seguente mostra i diversi prodotti ricombinanti che sono possibili.
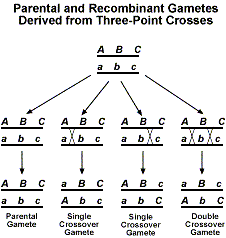
Ora se dovessimo eseguire un testcross con F1, ci aspetteremmo un rapporto 1:1:1:1:1:1:1:1:1:1:1. Come per le analisi a due punti descritte sopra, la deviazione da questo rapporto atteso indica che si sta verificando il linkage.Il modo migliore per familiarizzare con l’analisi dei dati di testcross a tre punti è quello di passare attraverso un esempio. Useremo l’esempio arbitrario dei geni A, B e C. Prima facciamo un incrocio tra individui che sono AABBCC e aabbcc. Poi il gene F1 viene incrociato con un individuo che è aabbcc. Useremo i seguenti dati per determinare l’ordine dei geni e le distanze di collegamento. Come per i dati a due punti, considereremo la composizione dei gameti F1.
| Genotipo | Osservato | Tipo di gamete | |
|---|---|---|---|
| ABC | Parentale | ||
| abc | Parentale | ||
| AbC | Single-crossover tra i geni C e B | ||
| aBc | Single-crossover tra i geni C e B | ||
| ABc | Double-crossover | ||
| abC | Doppio crossover | ||
| Abc | Crossover singolo tra i geni A e C | ||
| aBC | Single-crossover tra i geni A e C | ||
| Totale |
Il modo migliore per risolvere questi problemi è sviluppare un approccio sistematico.In primo luogo, determinare quali dei genotipi sono i genotipi parentali.I genotipi trovati più frequentemente sono i genotipi parentali.Dalla tabella è chiaro che i genotipi ABC e abc erano i genotipi parentali.
In seguito dobbiamo determinare l’ordine dei geni. Una volta determinati i genotipi dei genitori, usiamo questa informazione insieme a quella ottenuta dal doppio incrocio. I gameti del doppio incrocio sono sempre nella frequenza più bassa. Dalla tabella i genotipi ABc e abC sono nella frequenza più bassa. Il prossimo punto importante è che un evento di doppio incrocio sposta l’allele centrale da un cromatide sorella all’altro. Questo pone effettivamente l’allele non parentale del gene centrale su un cromosoma con gli alleli parentali dei due geni laterali. Possiamo vedere dalla tabella che il gene C deve essere al centro perché l’allele C recessivo è ora sullo stesso cromosoma degli alleli A e B, e l’allele C dominante è sullo stesso cromosoma degli alleli A e B recessivi.
Ora che sappiamo che l’ordine del gene è ACB, possiamo determinare le distanze di collegamento tra A e C, e C e B. La distanza di collegamento è calcolata dividendo il numero totale di gameti ricombinanti per il numero totale di gameti. Questo è lo stesso approccio che abbiamo usato con le analisi a due punti che abbiamo eseguito prima. Ciò che è diverso è che ora dobbiamo considerare anche gli eventi di doppio crossover. Per questi calcoli includiamo questi doppi incroci nei calcoli di entrambe le distanze di intervallo.
Quindi la distanza tra i geni A e C è 17,9 cM, e la distanza tra C e Bis 7,0 cM.
Ora proviamo un problema di Drosophila, applicando i principi che abbiamo usato nell’esempio precedente. La seguente tabella fornisce i risultati che analizzeremo.
| Genotipo | Osservato | Tipo di gamete | |
|---|---|---|---|
| v cv+ ct+ | Parentale | ||
| v+ cv ct | Parentale | ||
| v cv ct+ | Single-crossover tra i geni ct e cv | ||
| v+ cv+ ct | Single-crossover tra i geni ct e cv | ||
| v cv ct | Single-crossover tra i geni v e ct | ||
| v+ cv+ ct+ | Single-crossover tra i geni v e ct | ||
| v cv+ ct | Doppio-crossover | ||
| v+ cv ct+ | Double-crossover | ||
| Totale |
Passo 1: Determinare i genotipi dei genitori.
I genotipi più abbondanti sono i genotipi parentali. Questi genotipi sono v cv+ ct+ e v+ cv ct.Ciò che è diverso dal nostro primo incrocio a tre punti è che un genitore non contiene tutti gli alleli dominanti e l’altro tutti i recessivi.
Step 2: Determinare l’ordine del gene
Per determinare l’ordine del gene, abbiamo bisogno dei genotipi dei genitori e dei genotipi del doppio incrocio Come abbiamo detto sopra, i genotipi meno frequenti sono quelli del doppio incrocio. Questi genotipi sono v cv cv+ ct e v+ cv ct+. Da queste informazioni possiamo determinare l’ordine ponendoci la domanda: nei genotipi a doppio incrocio, quale allele parentale non è associato ai due alleli parentali con cui era associato nell’incrocio parentale originale. Dal primo doppio incrocio, v cv+ ct, l’allele ct è associato agli alleli v ecv+, due alleli con cui non era associato nell’incrocio originale. Pertanto, ct è nel mezzo, e l’ordine dei geni è v ct cv.
Step 3: Determinazione delle distanze di linkage.
-
Calcolo della distanza v – ct. Questa distanza è derivata come segue: 100*((89+94+3+5)/1448) = 13,2 cM
-
calcolo della distanza ct – cv. Questa distanza è derivata come segue: 100*((45+40+3+5)/1448) = 6,4 cM
Passo 4. Disegnare la mappa.

Gli incroci a tre punti permettono anche di misurare l’interferenza (I) tra gli eventi di crossover in una data regione di un cromosoma. Il concetto è che dati specifici tassi di ricombinazione in due intervalli cromosomici adiacenti, il tasso di doppio crossover in questa regione dovrebbe essere uguale al prodotto dei singoli crossover. Nell’esempio v ct cv descritto sopra, la frequenza di ricombinazione era 0,132 tra i geni v e ct, e la frequenza di ricombinazione tra ct e cv era 0,064. Pertanto, ci aspetteremmo lo 0,84% di ricombinanti doppi. Con una dimensione del campione di 1448, questo ammonterebbe a 12 doppi ricombinanti. In realtà abbiamo rilevato solo 8.
Per misurare l’interferenza, calcoliamo prima il coefficiente di coincidenza (c.o.c.) che è il rapporto tra i ricombinanti doppi osservati e attesi. L’interferenza viene quindi calcolata come 1 – c.o.c. La formula è la seguente:
![]()
Per i dati v ct cvdata, il valore di interferenza è 33%.
La maggior parte delle volte, i valori di interferenza cadono tra 0 e 1. Valori inferiori a uno indicano che l’interferenza si verifica in questa regione del cromosoma.