Kapcsolódási távolság és génrend levezetése hárompontos keresztezésekből
Egy harmadik gén hozzáadásával már többféle keresztezési túlterméket kaphatunk. A következő ábra mutatja a lehetséges különböző rekombináns termékeket.
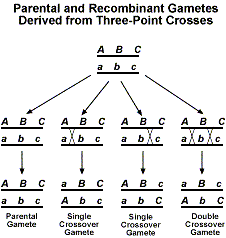
Ha most F1-gyel végeznénk egy tesztkeresztezést, akkor 1:1:1:1:1:1:1:1:1:1:1:1:1 arányt várnánk. A fent leírt kétpontos elemzésekhez hasonlóan az ettől a várt aránytól való eltérés azt jelzi, hogy kapcsolódás történik.A hárompontos tesztkeresztezési adatok elemzésével úgy ismerkedhetünk meg a legjobban, ha végigmegyünk egy példán. Az A, B és C gének tetszőleges példáját fogjuk használni. Először keresztezést készítünk AABBCC és aabbcc egyedek között. Ezután az F1-et tesztkeresztezzük egy olyan egyeddel, amely aabbcc. A következő adatokat fogjuk használni a génsorrend és a kapcsolási távolságok meghatározásához.A kétpontos adatokhoz hasonlóan az F1 ivarösszetételt fogjuk figyelembe venni.
| Genotípus | Megfigyelt | Ivarsejt típus | |
|---|---|---|---|
| ABC | Szülői | ||
| abc | Szülői | ||
| AbC | Single-crossover a C és B gének között | ||
| aBc | Single-crossover a C és B gének között | ||
| ABc | Double-crossover | ||
| abC | Double-crossover | ||
| Abc | Single-crossover A és C gének között | ||
| aBC | Single-crossover A és C gének között | ||
| Összesen |
A problémák megoldásának legjobb módja egy szisztematikus megközelítés kidolgozása.Először is meg kell határozni, hogy a genotípusok közül melyek a szülői genotípusok.A leggyakrabban talált genotípusok a szülői genotípusok.A táblázatból egyértelműen kiderül, hogy az ABC és abc genotípusok voltak a szülői genotípusok.
A következőkben a gének sorrendjét kell meghatározni. Miután meghatároztuk a szülői genotípusokat, ezt az információt használjuk fel a kettős keresztezésből nyert információval együtt. A kettős keresztezésű ivarsejtek mindig a legalacsonyabb gyakoriságúak. A táblázatból az ABcés abC genotípusok a legalacsonyabb gyakoriságúak. A következő fontos szempont az, hogy a kettős kereszteződéses esemény a középső allélt az egyik testvérkromatidáról a másikra helyezi át. Ezáltal a középső gén nem szülői allélja ténylegesen egy kromoszómára kerül a két flankáló gén szülői alléljával. A táblázatból láthatjuk, hogy a C génnek középen kell lennie, mert a recesszív c allél most ugyanazon a kromoszómán van, mint az A és B allél, és a dominánsC allél ugyanazon a kromoszómán van, mint a recesszív a és gömballél.
Most, hogy tudjuk, hogy a génsorrend ACB, meg tudjuk határozni az A és C, valamint a C és B közötti kapcsolási távolságokat. A kapcsolási távolságot úgy számítjuk ki, hogy a rekombináns ivarsejtek teljes számát elosztjuk az ivarsejtek teljes számával. Ez ugyanaz a megközelítés, amelyet a korábban elvégzett kétpontos elemzéseknél használtunk. A különbség az, hogy most már a kettős keresztezési eseményeket is figyelembe kell vennünk. Ezeknél a számításoknál ezeket a kettős kereszteződéseket is figyelembe vesszük mindkét intervallumtávolság számításánál.
Az A és C gének közötti távolság tehát 17,9 cM, a C és Bis közötti távolság pedig 7,0 cM .
Most próbáljuk ki a Drosophila problémáját, a fenti példában használt elvek alkalmazásával. A következő táblázat tartalmazza az eredményeket, amelyeket elemezni fogunk.
| genotípus | megfigyelt | ivarsejt típus | |
|---|---|---|---|
| v cv+ ct+ | Szülői | ||
| v+ cv ct | Szülői | ||
| v cv ct+ | Single-ct és cv gének keresztezése | ||
| v+ cv+ ct | Single-crossover a ct és cv gének között | ||
| v cv ct | Single-crossover a v és ct gének között | ||
| v+ cv+ ct+ | Single-crossover a v és ct gének között | ||
| v cv+ ct | Dupla-crossover | ||
| v+ cv ct+ | Double-crossover | ||
| Összesen |
1. lépés: A szülői genotípusok meghatározása.
A leggyakoribb genotípusok a szülői típusok. Ezek a genotípusokv cv+ ct+ és v+ cv ct.Az első hárompontos keresztezésünkhöz képest az a különbség, hogy az egyik szülőnem tartalmazza az összes domináns allélt, a másik pedig az összes recesszív allélt.
2. lépés: A génsorrend meghatározása
A génsorrend meghatározásához szükségünk van a szülői genotípusokra, valamint adupla keresztezéses genotípusokra Mint már említettük, a legritkább genotípusok a dupla keresztezéses genotípusok. Ezek a géntípusokv cv+ ct és v+ cv ct+.Ebből az információból a sorrendet a következő kérdéssel határozhatjuk meg:A kettős keresztezés genotípusaiban melyik szülői allél nem társul azzal a két szülői alléllal, amellyel az eredeti szülői keresztezésben társult. Az első kettős kereszteződésből, v cv+ ct,a ct allél társul a v és acv+ alléllal, két alléllal, amelyekkel nem társult az eredeti kereszteződésben. Ezért a ct középen van, és a génrend v ct cv.
3. lépés: A kapcsolási távolságok meghatározása.
-
v – ct távolságkalkuláció. Ezt a távolságot a következőképpen származtatjuk: 100*((89+94+3+5)/1448) = 13,2 cM
-
ct – cv távolság számítása. Ezt a távolságot a következőképpen származtatjuk: 100*((45+40+3+5)/1448) = 6,4 cM
4. lépés. Rajzoljuk meg a térképet.

A hárompontos keresztezések lehetővé teszik a kromoszóma egy adott régióján belüli keresztezési események közötti interferencia(I) mérését is.Pontosabban, a kettős keresztezés mennyisége jelzi, ha interferencia fordul elő. A koncepció az, hogy adott rekombinációs arányok mellett két szomszédos kromoszómaintervallumban a kettős kereszteződések arányának ebben a régióban meg kell egyeznie az egyszeres kereszteződések szorzatával. A fent leírt v ct cv példában a rekombinációsfrekvencia 0,132 volt a v és ct gének között, és a rekombinációsfrekvencia a ct és cv gének között 0,064 volt. Ezért 0,84% kettős rekombinációt várnánk. Egy 1448 fős mintanagyság mellett ez 12 kettős rekombinánst jelentene. Valójában csak 8-at észleltünk.
Az interferencia méréséhez először kiszámítjuk a koincidencia együtthatót (c.o.c.), amely a megfigyelt és a várt kettős rekombinánsok aránya. Az interferenciát ezután 1 – c.o.c.-ként számítjuk ki.A képlet a következő:
![]()
A v ct cvdata esetében az interferencia értéke 33%.
A leggyakrabban az interferencia értéke 0 és 1 közé esik. Az egynél kisebb értékek azt jelzik, hogy a kromoszómának ebben a régiójában interferencia lép fel.