Dérivation de la distance de liaison et de l’ordre des gènes à partir de croisements à trois points
En ajoutant un troisième gène, nous avons maintenant plusieurs types différents de produits de recombinaison qui peuvent être obtenus. La figure suivante montre les différents produits recombinants possibles.
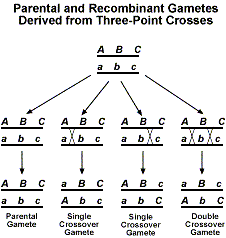
Maintenant, si nous devions effectuer un testcross avec F1, nous nous attendrions à un rapport 1:1:1:1:1:1:1:1:1. Comme pour les analyses à deux points décrites ci-dessus, la déviation de ce rapport attendu indique qu’il y a liaison.La meilleure façon de se familiariser avec l’analyse des données de testcross à trois points est de prendre un exemple. Nous utiliserons l’exemple arbitraire des gènes A, B et C. Nous faisons d’abord un croisement entre des individus qui sont AABBCC et aabbcc. Ensuite, le gène F1 est croisé avec un individu qui est aabbcc. Nous utiliserons les données suivantes pour déterminer l’ordre des gènes et les distances de liaison.Comme pour les données à deux points, nous considérerons la composition des gamètes F1.
| Génotype | Observé | Type de gamète |
|---|---|---|
| ABC | . Parental | |
| abc | Parental | |
| AbC | Simple-croisement entre les gènes C et B | |
| aBc | Croisement simple entre les gènes C et B | |
| ABc | Double-croisement | |
| abC | Double-croisement | |
| Abc | Simple-croisement entre les gènes A et C | |
| aBC | Simple-crossover entre les gènes A et C | |
| Total |
La meilleure façon de résoudre ces problèmes est de développer une approche systématique.Tout d’abord, déterminer quels sont les génotypes parentaux.Les génotypes trouvés le plus fréquemment sont les génotypes parentaux.D’après le tableau, il est clair que les génotypes ABC et abc étaient les génotypes parentaux.
Puis nous devons déterminer l’ordre des gènes. Une fois que nous avons déterminé les génotypes parentaux, nous utilisons cette information avec l’information obtenue par le double croisement. Les gamètes du double croisement ont toujours la fréquence la plus basse. D’après le tableau, les génotypes ABc et abC ont la fréquence la plus basse. Le point important suivant est qu’un événement de double croisement déplace l’allèle central d’une chromatide sœur à l’autre. Cela place effectivement l’allèle non parental du gène central sur un chromosome avec les allèles parentaux des deux gènes adjacents. Nous pouvons voir dans le tableau que le gène C doit être au milieu parce que l’allèle récessif c est maintenant sur le même chromosome que les allèles A et B, et l’allèle dominantC est sur le même chromosome que les allèles récessifs a et balle.
Maintenant que nous savons que l’ordre des gènes est ACB, nous pouvons passer à la détermination des distances de liaison entre A et C, et C etB. La distance de liaison est calculée en divisant le nombre total de gamètes recombinants par le nombre total de gamètes. Il s’agit de la même approche que celle utilisée pour les analyses en deux points que nous avons effectuées plus tôt. Ce qui est différent, c’est que nous devons maintenant tenir compte également des événements de double croisement. Pour ces calculs, nous incluons ces doubles croisements dans les calculs des deux distances d’intervalle.
Donc la distance entre les gènes A et C est de 17,9 cM, et la distance entre C et Bis de 7,0 cM .
Tentons maintenant un problème à partir de la drosophile, en appliquant les principes que nous avons utilisés dans l’exemple ci-dessus. Le tableau suivant donne les résultats que nous allons analyser.
| Génotype | Observé | Type de gamète |
|---|---|---|
| v cv+ ct+ | . Parental | |
| v+ cv ct | Parental | |
| v cv ct+ | Simple-croisement entre les gènes ct et cv | |
| v+ cv+ ct | Simple-crossover entre les gènes ct et cv | |
| v cv ct | Single-crossover entre les gènes v et ct | |
| v+ cv+ ct+ | Single-crossover entre les gènes v et ct | |
| v cv+ ct | Double-crossover | |
| v+ cv ct+ | Double-crossover | |
| Total |
Étape 1 : Déterminer les génotypes parentaux.
Les génotypes les plus abondants sont les types partenariaux. Ces génotypes sont v cv+ ct+ et v+ cv ct.Ce qui est différent de notre premier croisement à trois points, c’est qu’un parent ne contenait pas tous les allèles dominants et l’autre tous les allèles récessifs.
Étape 2 : Déterminer l’ordre des gènes
Pour déterminer l’ordre des gènes, nous avons besoin des génotypes parentaux ainsi que des génotypes de double croisement Comme nous l’avons mentionné plus haut, les génotypes les moins fréquents sont les génotypes de double croisement. À partir de ces informations, nous pouvons déterminer l’ordre en posant la question suivante : dans les génotypes doublement croisés, quel est l’allèle parental qui n’est pas associé aux deux allèles parentaux avec lesquels il était associé dans le croisement parental initial. Dans le premier double croisement, v cv+ ct, l’allèle ct est associé aux allèles v et cv+, deux allèles auxquels il n’était pas associé dans le croisement original. Par conséquent, ct est au milieu, et l’ordre des gènes est v ct cv.
Etape 3 : Détermination des distances de liaison.
-
Caluculation de la distance v – ct. Cette distance est calculée comme suit : 100*((89+94+3+5)/1448) = 13,2 cM
-
Calcul de la distance ct – cv. Cette distance est calculée comme suit : 100*((45+40+3+5)/1448) = 6,4 cM
Étape 4. Dessiner la carte.

Les croisements à trois points permettent également de mesurer l’interférence (I) entre les événements de croisement dans une région donnée d’un chromosome.Plus précisément, la quantité de double croisement donne une indication si une interférence se produit. Le concept est le suivant : étant donné des taux de recombinaison spécifiques dans deux intervalles chromosomiques adjacents, le taux de double croisement dans cette région devrait être égal au produit des croisements simples. Dans l’exemple v ct cv décrit ci-dessus, la fréquence de recombinaison était de 0,132 entre les gènes v et ct, et la fréquence de recombinaison entre ct et cv était de 0,064. Par conséquent, nous nous attendons à 0,84% de doubles recombinants. Avec une taille d’échantillon de 1448, cela équivaudrait à 12 doubles recombinants. Nous n’en avons en fait détecté que 8.
Pour mesurer l’interférence, nous calculons d’abord le coefficient d’coïncidence (c.o.c.) qui est le rapport entre les recombinants doubles observés et attendus. L’interférence est ensuite calculée comme 1 – c.o.c.La formule est la suivante :
![]()
Pour les données v ct cvdata, la valeur d’interférence est de 33%.
Le plus souvent, les valeurs d’interférence se situent entre 0 et 1. Les valeurs inférieures à un indiquent que l’interférence se produit dans cette région du chromosome.